18 Valores faltantes
18.1 Introdução
Você já aprendeu o básico sobre valores faltantes (missing values) no início deste livro. Você os viu pela primeira vez no Capítulo 1, onde resultaram em um aviso ao criar um gráfico, bem como na Seção 3.5.2, onde interferiram no cálculo de estatísticas sumarizadas, e aprendeu sobre sua natureza de propagação e como verificar sua presença na Seção 12.2.2. Agora voltaremos a eles com mais profundidade, para que você possa aprender mais detalhes.
Começaremos discutindo algumas ferramentas gerais para trabalhar com valores faltantes registrados como NA (do inglês Not Available - “não disponível”). Em seguida, exploraremos a ideia de valores faltantes implícitos, ou seja, valores que estão simplesmente ausentes em seus dados, e mostraremos algumas ferramentas que você pode usar para torná-los explícitos. Terminaremos com uma discussão relacionada sobre grupos vazios, causados por níveis de fatores que não aparecem nos dados.
18.1.1 Pré-requisitos
As funções para trabalhar com dados ausentes vêm principalmente dos pacotes dplyr e tidyr, que são membros principais do tidyverse.
18.2 Valores faltantes explícitos
Para começar, vamos explorar algumas ferramentas úteis para criar ou eliminar valores explícitos ausentes, ou seja, células onde você vê um NA.
18.2.1 Última observação levada adiante
Um uso comum de valores faltantes (missing values) é como conveniência na entrada de dados. Quando os dados são inseridos manualmente, os valores ausentes às vezes indicam que o valor da linha anterior foi repetido (levado adiante):
tratamento <- tribble(
~pessoa, ~tratamento, ~resposta,
"Derrick Whitmore", 1, 7,
NA, 2, 10,
NA, 3, NA,
"Katherine Burke", 1, 4
)Você pode preencher esses valores ausentes com a função tidyr::fill(). Funciona como a função select(), pegando um conjunto de colunas como argumento:
tratamento |>
fill(everything())
#> # A tibble: 4 × 3
#> pessoa tratamento resposta
#> <chr> <dbl> <dbl>
#> 1 Derrick Whitmore 1 7
#> 2 Derrick Whitmore 2 10
#> 3 Derrick Whitmore 3 10
#> 4 Katherine Burke 1 4Este tratamento às vezes é chamado de “última observação realizada” (last observation carried forward) ou locf, abreviadamente. Você pode usar o argumento .direction para preencher valores ausentes que foram gerados de maneiras mais exóticas.
18.2.2 Valores fixos
Algumas vezes, os valores ausentes representam algum valor fixo e conhecido, mais comumente 0. Você pode usar a dplyr::coalesce() para substituí-los:
Às vezes você encontrará o problema oposto, onde algum valor concreto na verdade representa um valor ausente. Isso normalmente surge em dados gerados por softwares mais antigos que não possuem uma maneira adequada de representar valores ausentes; portanto, deve-se usar algum valor especial como 99 ou -999.
Se possível, lide com isso ao ler os dados, por exemplo, usando o argumento na para readr::read_csv(), por exemplo, read_csv(caminho, na = "99"). Se você descobrir o problema mais tarde, ou se sua fonte de dados não fornecer uma maneira de lidar com isso na leitura, você pode usar dplyr::na_if():
18.2.3 NaN
Antes de continuarmos, há um tipo especial de valor ausente que você encontrará de tempos em tempos: um NaN (pronuncia-se “nan”) ou em inglês, not a number (em português, seria como “não é um número”). Não é tão importante saber sobre ele, já que geralmente se comporta como NA:
Se você tiver um caso raro e precisar distinguir um NA de um NaN, você pode usar is.nan(x).
Você geralmente encontrará um NaN ao realizar uma operação matemática que tem um resultado indeterminado:
0 / 0
#> [1] NaN
0 * Inf
#> [1] NaN
Inf - Inf
#> [1] NaN
sqrt(-1)
#> Warning in sqrt(-1): NaNs produced
#> [1] NaN18.3 Valores faltantes implícitos
Até agora falamos sobre valores que estão explicitamente ausentes, ou seja, você pode ver um NA em seus dados. Mas os valores também podem estar implicitamente ausentes, se uma linha inteira de dados estiver simplesmente ausente dos dados. Vamos ilustrar a diferença com um conjunto de dados simples que registra o preço de algumas ações a cada trimestre:
Este conjunto de dados tem duas observações faltantes:
O
precono quarto trimestre de 2020 está explicitamente ausente, porque o seu valor éNA.O
preçopara o primeiro trimestre de 2021 está implicitamente ausente, porque simplesmente não aparece no conjunto de dados.
Uma maneira de pensar sobre a diferença é como este diálogo meio filosófico:
Um valor ausente explícito é a presença de uma ausência.
Um valor ausente implícito é a ausência de presença.
Às vezes, você deseja tornar explícitos os valores ausentes implícitos para ter algo físico com o qual trabalhar. Em outros casos, valores faltantes explícitos são impostos pela estrutura dos dados e você deseja se livrar deles. As seções a seguir discutem algumas ferramentas para alternar entre ausências implícitas e explícitas.
18.3.1 Pivotagem
Você já viu uma ferramenta que pode tornar explícitas as ausências implícitas e vice-versa: pivotagem. Deixar os dados mais largos (com mais colunas) pode tornar explícitos os valores ausentes implícitos porque cada combinação de linhas e novas colunas deve ter algum valor. Por exemplo, se pivotarmos acoes para colocar o trimestre nas colunas, ambos os valores faltantes se tornarão explícitos:
acoes |>
pivot_wider(
names_from = trimestre,
values_from = preco
)
#> # A tibble: 2 × 5
#> ano `1` `2` `3` `4`
#> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 2020 1.88 0.59 0.35 NA
#> 2 2021 NA 0.92 0.17 2.66Por padrão, tornar os dados mais longos preserva os valores ausentes explícitos, mas se eles forem valores estruturalmente ausentes que só existem porque os dados não estão organizados, você pode eliminá-los (torná-los implícitos) definindo values_drop_na = TRUE. Veja os exemplos na Seção 5.2 para mais detalhes.
18.3.2 Completar
A função tidyr::complete() permite gerar valores faltantes explícitos, fornecendo um conjunto de variáveis que definem a combinação de linhas que devem existir. Por exemplo, sabemos que todas as combinações de ano e trimestre devem existir nos dados de acoes:
acoes |>
complete(ano, trimestre)
#> # A tibble: 8 × 3
#> ano trimestre preco
#> <dbl> <dbl> <dbl>
#> 1 2020 1 1.88
#> 2 2020 2 0.59
#> 3 2020 3 0.35
#> 4 2020 4 NA
#> 5 2021 1 NA
#> 6 2021 2 0.92
#> # ℹ 2 more rowsNormalmente, você chamará complete() com nomes de variáveis existentes, preenchendo as combinações que faltam. No entanto, às vezes as variáveis individuais estão incompletas, então você pode fornecer seus próprios dados. Por exemplo, você pode saber que o conjunto de dados acoes deve ser executado de 2019 a 2021, então você pode fornecer explicitamente esses valores para ano:
acoes |>
complete(ano = 2019:2021, trimestre)
#> # A tibble: 12 × 3
#> ano trimestre preco
#> <dbl> <dbl> <dbl>
#> 1 2019 1 NA
#> 2 2019 2 NA
#> 3 2019 3 NA
#> 4 2019 4 NA
#> 5 2020 1 1.88
#> 6 2020 2 0.59
#> # ℹ 6 more rowsSe o intervalo de uma variável estiver correto, mas nem todos os valores estiverem presentes, você pode usar a full_seq(x, 1) para gerar todos os valores de min(x) a max(x) espaçados por 1.
Em alguns casos, o conjunto completo de observações não pode ser gerado por uma simples combinação de variáveis. Nesse caso, você pode fazer manualmente o que complete() faz por você: criar um dataframe que contenha todas as linhas que deveriam existir (usando qualquer combinação de técnicas necessárias) e, em seguida, combiná-lo com seu conjunto de dados original com dplyr::full_join().
18.3.3 Uniões (joins)
Isso nos leva a outra forma importante de revelar observações implicitamente ausentes: uniões (joins). Você aprenderá mais sobre uniões na Capítulo 19, mas gostaríamos de mencioná-las rapidamente aqui, já que muitas vezes você só pode saber que valores estão faltando em um conjunto de dados quando você o compara com outro.
dplyr::anti_join(x, y) é uma função particularmente útil aqui porque seleciona apenas as linhas em x que não têm correspondência em y. Por exemplo, podemos usar dois anti_join() para revelar que faltam informações de quatro aeroportos e 722 aviões mencionados em voos:
library(dados)
voos |>
distinct(codigo_aeroporto = destino) |>
anti_join(aeroportos)
#> Joining with `by = join_by(codigo_aeroporto)`
#> # A tibble: 4 × 1
#> codigo_aeroporto
#> <chr>
#> 1 BQN
#> 2 SJU
#> 3 STT
#> 4 PSE
voos |>
distinct(codigo_cauda) |>
anti_join(avioes)
#> Joining with `by = join_by(codigo_cauda)`
#> # A tibble: 722 × 1
#> codigo_cauda
#> <chr>
#> 1 N3ALAA
#> 2 N3DUAA
#> 3 N542MQ
#> 4 N730MQ
#> 5 N9EAMQ
#> 6 N532UA
#> # ℹ 716 more rows18.3.4 Exercícios
- Você consegue encontrar alguma relação entre a companhia_aerea e as linhas que parecem estar faltando em
avioes?
18.4 Fatores e grupos vazios
Um último tipo de ausência é a de grupo vazio, um grupo que não contém nenhuma observação, que pode surgir ao trabalhar com fatores (factors). Por exemplo, imagine que temos um conjunto de dados que contém algumas informações de saúde sobre pessoas:
E queremos contar o número de fumantes com a função dplyr::count():
saude |> count(fumante)
#> # A tibble: 1 × 2
#> fumante n
#> <fct> <int>
#> 1 nao 5Este conjunto de dados contém apenas não fumantes, mas sabemos que existem fumantes; o grupo de não fumantes está vazio. Podemos solicitar count() para manter todos os grupos, mesmo aqueles não vistos nos dados usando .drop = FALSE:
saude |> count(fumante, .drop = FALSE)
#> # A tibble: 2 × 2
#> fumante n
#> <fct> <int>
#> 1 sim 0
#> 2 nao 5O mesmo princípio se aplica aos eixos discretos (discrete axis) do ggplot2, que também eliminarão níveis que não possuem nenhum valor. Você pode forçá-los a serem exibidos fornecendo drop = FALSE ao eixo discreto apropriado:
ggplot(saude, aes(x = fumante)) +
geom_bar() +
scale_x_discrete()
ggplot(saude, aes(x = fumante)) +
geom_bar() +
scale_x_discrete(drop = FALSE)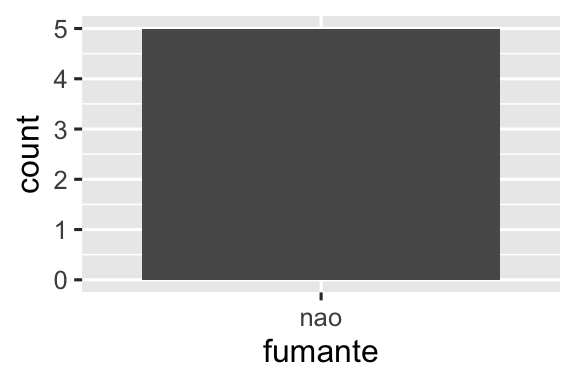
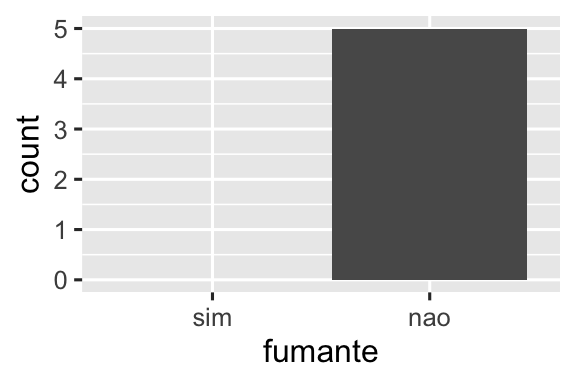
O mesmo problema surge de forma mais geral com dplyr::group_by(). E novamente você pode usar .drop = FALSE para preservar todos os níveis dos fatores:
saude |>
group_by(fumante, .drop = FALSE) |>
summarize(
n = n(),
idade_media = mean(idade),
idade_min = min(idade),
idade_max = max(idade),
idade_desvpad = sd(idade)
)
#> # A tibble: 2 × 6
#> fumante n idade_media idade_min idade_max idade_desvpad
#> <fct> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 sim 0 NaN Inf -Inf NA
#> 2 nao 5 60 34 88 21.6Obtemos alguns resultados interessantes aqui porque ao sumarizar um grupo vazio, as funções de sumarização são aplicadas a vetores de comprimento zero. Há uma distinção importante entre vetores vazios, que têm comprimento 0, e valores ausentes, cada um dos quais tem comprimento 1.
Todas as funções de sumarização funcionam com vetores de comprimento zero, mas podem retornar resultados surpreendentes à primeira vista. Aqui vemos mean(idade) retornando NaN porque mean(idade) = sum(age)/length(idade) que aqui é 0/0. max() e min() retornam -Inf e Inf para vetores vazios, então se você combinar os resultados com um vetor não vazio de novos dados e recalcular, você obterá o mínimo ou máximo dos novos dados1.
Às vezes, uma abordagem mais simples é realizar a sumarização e então tornar explícitas as faltas implícitas com complete().
saude |>
group_by(fumante) |>
summarize(
n = n(),
idade_media = mean(idade),
idade_min = min(idade),
idade_max = max(idade),
idade_desvpad = sd(idade)
) |>
complete(fumante)
#> # A tibble: 2 × 6
#> fumante n idade_media idade_min idade_max idade_desvpad
#> <fct> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 sim NA NA NA NA NA
#> 2 nao 5 60 34 88 21.6A principal desvantagem desta abordagem é que você obtém um NA para a contagem, mesmo sabendo que deveria ser zero.
18.5 Resumo
Valores faltantes são estranhos! Às vezes eles são registrados como um NA explícito, mas outras vezes você só os percebe pela sua ausência. Este capítulo forneceu algumas ferramentas para trabalhar com valores faltantes explícitos, ferramentas para descobrir valores faltantes implícitos e discutiu algumas das maneiras pelas quais os implícitos podem se tornar explícitos e vice-versa.
No próximo capítulo, abordamos o capítulo final desta parte do livro: uniões. Isso é uma pequena mudança em relação aos capítulos até agora porque discutiremos ferramentas que funcionam com data frames como um todo, não algo que você coloca dentro de um data frame.
Em outras palavras,
min(c(x, y))é sempre igual amin(min(x), min(y)).↩︎