5 Organização de dados (data tidying)
5.1 Introdução
“Todas as famílias felizes se parecem, cada família infeliz é infeliz à sua maneira.”
— Leo Tolstoy
“Todos os conjuntos de dados organizados se parecem, cada conjunto de dados bagunçados são bagunçados à sua maneira.”
— Hadley Wickham
Neste capítulo, você vai aprender uma forma consistente de organizar seus dados no R utilizando um sistema chamado tidy data. Colocar seus dados nesse formato requer alguma preparação, mas esse é um trabalho que se paga ao longo prazo. Uma vez que você tenha os dados organizados (tidy) e as ferramentas do pacote tidyverse, você irá gastar muito menos tempo transformando-os entre uma representação e outra, permitindo que você gaste mais tempo nas questões que realmente te importam.
Neste capítulo, você primeiro vai aprender sobre a definição do formato tidy e vê-lo sendo aplicado em um conjunto de dados de exemplo. Em seguida, você vai mergulhar na principal ferramenta que utilizará para organizar os dados: a pivotagem (pivoting). Pivotar lhe permite mudar a forma dos dados sem modificar nenhum de seus valores.
5.1.1 Pré-requisitos
Neste capítulo, vamos focar no tidyr, um pacote que disponibliza uma variedade de ferramentas para te ajudar a organizar seus conjuntos de dados bagunçados. tidyr faz parte do grupo de pacotes mais importantes do tidyverse (conhecidos como core tidyverse).
Deste capítulo em diante, vamos suprimir a mensagem de carregamento library(tidyverse).
5.2 Dados organizados (tidy data)
É possível representar os mesmos dados de diversas formas. O exemplo abaixo mostra os mesmos dados organizados de três formas diferentes. Cada conjunto de dados mostra os mesmos valores de quatro variáveis: país, ano, população, e o número de casos documentados de tuberculose. No entanto, cada conjunto de dados organiza os valores de uma forma diferente.
tabela1# A tibble: 6 × 4
pais ano casos populacao
<chr> <dbl> <dbl> <dbl>
1 Afeganistão 1999 745 19987071
2 Afeganistão 2000 2666 20595360
3 Brasil 1999 37737 172006362
4 Brasil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583tabela2# A tibble: 12 × 4
pais ano tipo contagem
<chr> <dbl> <chr> <dbl>
1 Afeganistão 1999 casos 745
2 Afeganistão 1999 população 19987071
3 Afeganistão 2000 casos 2666
4 Afeganistão 2000 população 20595360
5 Brasil 1999 casos 37737
6 Brasil 1999 população 172006362
7 Brasil 2000 casos 80488
8 Brasil 2000 população 174504898
9 China 1999 casos 212258
10 China 1999 população 1272915272
11 China 2000 casos 213766
12 China 2000 população 1280428583tabela3# A tibble: 6 × 3
pais ano taxa
<chr> <dbl> <chr>
1 Afeganistão 1999 745/19987071
2 Afeganistão 2000 2666/20595360
3 Brasil 1999 37737/172006362
4 Brasil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583Todas essas são representações dos mesmos dados. Mas nem todas são igualmente fáceis de se utilizar. Uma delas, tabela1, será muito mais fácil de trabalhar dentro do tidyverse porque está no formato tidy.
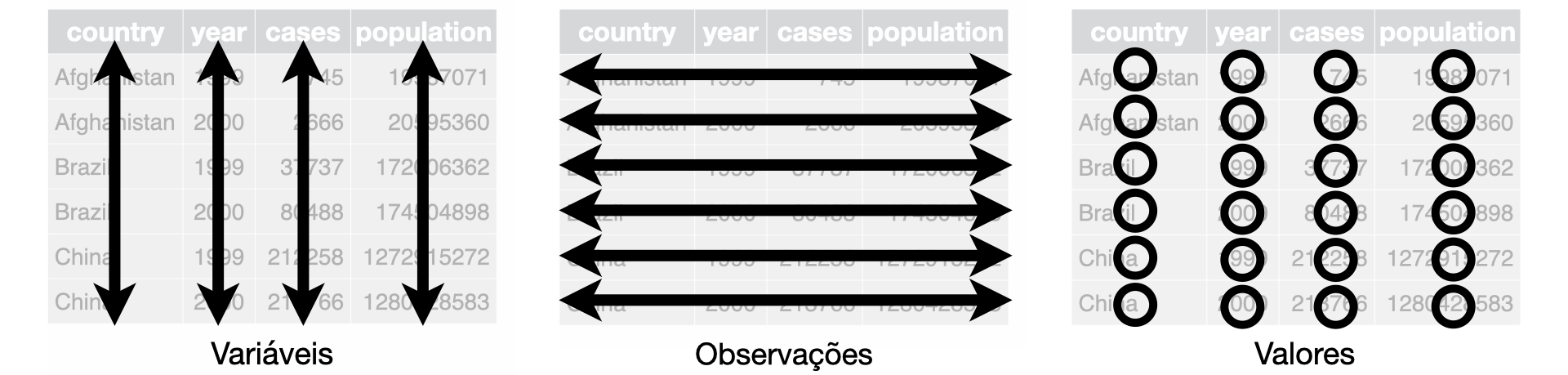
Existem três regras interrelacionadas que fazem com que um conjunto de dados seja considerado tidy:
- Cada variável é uma coluna; cada coluna é uma variável.
- Cada observação é uma linha; cada linha é uma observação.
- Cada valor é uma célula; cada célula é um único valor.
Figura 5.1 mostra essas regras visualmente.
Por que garantir que seus dados estão no formato tidy? Existem duas principais vantagens:
Há uma vantagem generalizada em escolher uma forma consistente de armazenar os dados. Se você tiver uma estrutura de dados consistente, é mais fácil compreender as ferramentas adequadas a ela, pois ela terá uma uniformidade intrínseca.
Há uma vantagem específica em colocar as variáveis em colunas porque isso permite que a natureza vetorial do R possa brilhar. Como você aprendeu em Seção 3.3.1 e Seção 3.5.2, a maioria das funções nativas do R trabalham com vetores de valores. Isso torna natural as transformações sobre dados no formato tidy.
dplyr, ggplot2, e todos os demais pacotes do tidyverse foram pensados para trabalhar com dados tidy. Aqui estão alguns pequenos exemplos mostrando como é possível trabalhar com tabela1.
# Calcular a taxa (_rate_) por 10.000
tabela1 |>
mutate(taxa = casos / populacao * 10000)# A tibble: 6 × 5
pais ano casos populacao taxa
<chr> <dbl> <dbl> <dbl> <dbl>
1 Afeganistão 1999 745 19987071 0.373
2 Afeganistão 2000 2666 20595360 1.29
3 Brasil 1999 37737 172006362 2.19
4 Brasil 2000 80488 174504898 4.61
5 China 1999 212258 1272915272 1.67
6 China 2000 213766 1280428583 1.67 # A tibble: 2 × 2
ano total_casos
<dbl> <dbl>
1 1999 250740
2 2000 296920# Visualizar mudanças ao longo do tempo
ggplot(tabela1, aes(x = ano, y = casos)) +
geom_line(aes(group = pais), color = "grey50") +
geom_point(aes(color = pais, shape = pais)) +
scale_x_continuous(breaks = c(1999, 2000)) # quebras (breaks) no eixo-x em 1999 e 2000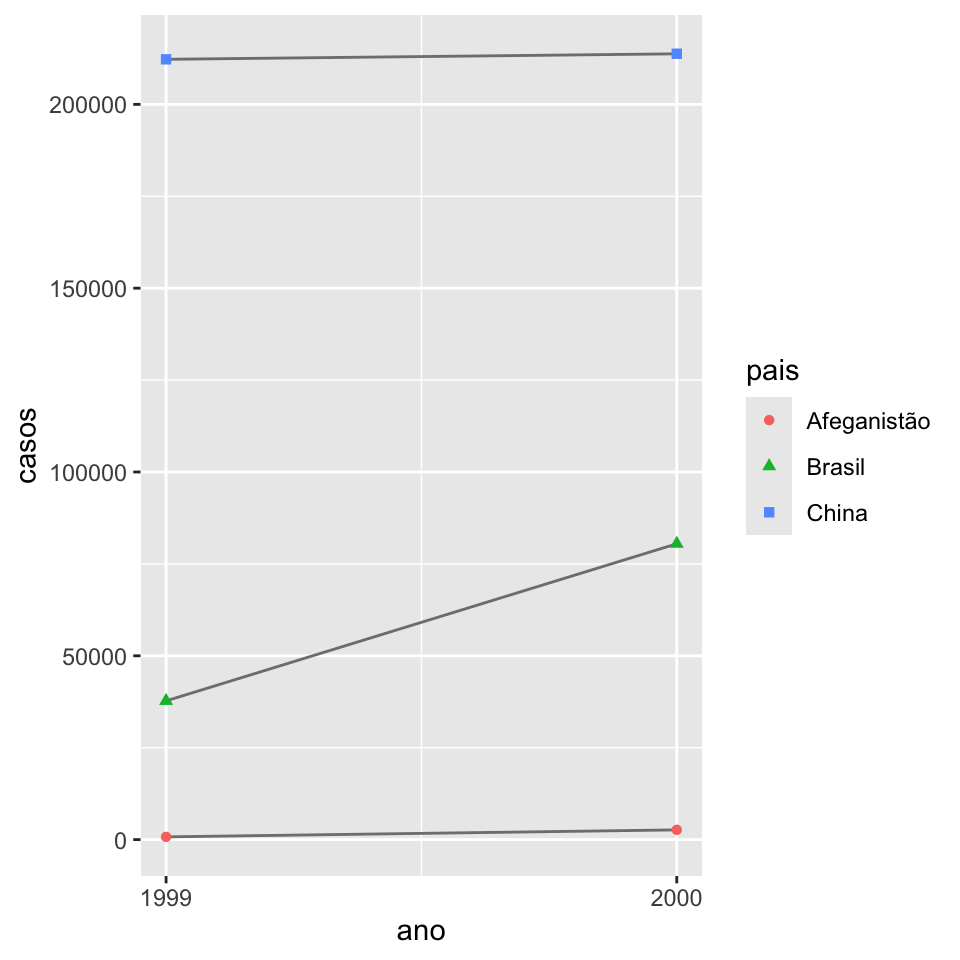
5.2.1 Exercícios
Para cada uma das tabelas do exemplo, descreva o que cada observação e cada coluna representa.
-
Faça um esboço do processo que você usaria para calcular
taxapara atabela2etabela3. Você precisará executar quatro operações:- Extrair o número de casos de tuberculose por país por ano.
- Extrair a população correspondente por país por ano.
- Dividir os casos pela população e multiplicar por 10000.
- Armazenar de volta no local apropriado.
Você ainda não aprendeu todas as funções necessárias para, de fato, executar essas operações, mas ainda assim você deve ser capaz de pensar sobre as transformações que você precisaria.
5.3 Alongando os dados
Os princípios do formato tidy parecem ser tão óbvios que você deve estar se perguntando se algum dia vai encontrar algum conjunto de dados que não esteja nesse formato. Infelizmente, porém, a maioria dos dados reais não estão nesse formato. Existem dois motivos principais:
Geralmente os dados estão organizados para facilitar algum outro objetivo além da análise. Por exemplo, é comum os dados estarem estruturados para facilitar a entrada de novos dados e não a análise dos mesmos.
A maioria das pessoas não está familiarizada com os princípios do formato tidy e é difícil derivá-lo espontaneamente a menos que já se tenha trabalhado com dados por muito tempo.
Isso significa que a maioria das análises reais vai demandar pelo menos alguma adaptação para o formato tidy. Você vai começar descobrindo quais são as variáveis e observações presentes. Algumas vezes isso é fácil, em outras você precisará consultar as pessoas que geraram os dados originalmente. Em seguida, você irá pivotar seus dados para o formato tidy, com as variáveis nas colunas e as observações nas linhas.
tidyr disponibiliza duas funções para pivotar os dados: pivot_longer() e pivot_wider(). Começaremos com pivot_longer() pois é o caso mais comum. Vamos nos aprofundar em alguns exemplos.
5.3.1 Dados nos nomes das colunas
O conjunto de dados top100musicas marca a posição das músicas na billboard no ano 2000:
top100musicas# A tibble: 317 × 79
artista musica data.adicionada semana1 semana2 semana3 semana4 semana5
<chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2 Pac Baby … 2000-02-26 87 82 72 77 87
2 2Ge+her The H… 2000-09-02 91 87 92 NA NA
3 3 Doors Down Krypt… 2000-04-08 81 70 68 67 66
4 3 Doors Down Loser 2000-10-21 76 76 72 69 67
5 504 Boyz Wobbl… 2000-04-15 57 34 25 17 17
6 98^0 Give … 2000-08-19 51 39 34 26 26
7 A*Teens Danci… 2000-07-08 97 97 96 95 100
8 Aaliyah I Don… 2000-01-29 84 62 51 41 38
9 Aaliyah Try A… 2000-03-18 59 53 38 28 21
10 Adams, Yolanda Open … 2000-08-26 76 76 74 69 68
# ℹ 307 more rows
# ℹ 71 more variables: semana6 <dbl>, semana7 <dbl>, semana8 <dbl>,
# semana9 <dbl>, semana10 <dbl>, semana11 <dbl>, semana12 <dbl>,
# semana13 <dbl>, semana14 <dbl>, semana15 <dbl>, semana16 <dbl>,
# semana17 <dbl>, semana18 <dbl>, semana19 <dbl>, semana20 <dbl>,
# semana21 <dbl>, semana22 <dbl>, semana23 <dbl>, semana24 <dbl>,
# semana25 <dbl>, semana26 <dbl>, semana27 <dbl>, semana28 <dbl>, …Nesse conjunto de dados, cada observação é uma música. As três primeiras colunas (artista, musica e data.adicionada) são variáveis que descrevem a música. Em seguida, temos 76 colunas (semana1-semana76) que descrevem a posição da música em cada semana1. Aqui, o nome das colunas é uma variável (a semana) e o valor da célula é outra (a posicao).
Para transformar esses dados em tidy, vamos usar pivot_longer():
top100musicas|>
pivot_longer(
cols = starts_with("semana"),
names_to = "semana",
values_to = "posicao"
)# A tibble: 24,092 × 5
artista musica data.adicionada semana posicao
<chr> <chr> <date> <chr> <dbl>
1 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana1 87
2 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana2 82
3 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana3 72
4 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana4 77
5 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana5 87
6 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana6 94
7 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana7 99
8 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana8 NA
9 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana9 NA
10 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana10 NA
# ℹ 24,082 more rowsNa função, são três argumentos principais, após os dados:
-
colsespecifica quais colunas precisarão ser pivotadas, ou seja, quais colunas ainda não são variáveis. Esse argumento usa a mesma sintaxe que a funçãoselect()então aqui poderíamos usar!c(artista, musica, data.adicionada)oustarts_with("semana"). -
names_tonomeia a variável armazenada nos nomes das colunas, nós nomeamos aquela variável comosemana. -
values_tonomeia a variável armazenada nos valores das células, nós nomeamos aquela variável comoposicao.
Note que os códigos "semana" e "posicao" estão entre aspas porque essas são novas variáveis que estamos criando, elas ainda não existem nos dados quando a gente faz a chamada à pivot_longer().
Agora voltaremos nossa atenção ao data frame resultante, em um formato mais longo. O que acontece se uma música fica no top 100 por menos que 76 semanas? Vejamos “Baby Don’t Cry” do 2 Pac, por exemplo. A saída acima sugere que ela ficou apenas 7 semanas no top 100, e todas as demais semanas foram preenchidas por valores ausentes. Esses NAs, na verdade, não representam observações desconhecidas; eles foram forçadas a existir pela estrutura do conjunto de dados 2, então podemos pedir para a pivot_longer() se livrar deles por meio do parâmetro values_drop_na = TRUE:
top100musicas|>
pivot_longer(
cols = starts_with("semana"),
names_to = "semana",
values_to = "posicao",
values_drop_na = TRUE
)# A tibble: 5,307 × 5
artista musica data.adicionada semana posicao
<chr> <chr> <date> <chr> <dbl>
1 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana1 87
2 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana2 82
3 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana3 72
4 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana4 77
5 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana5 87
6 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana6 94
7 2 Pac Baby Don't Cry (Keep... 2000-02-26 semana7 99
8 2Ge+her The Hardest Part Of ... 2000-09-02 semana1 91
9 2Ge+her The Hardest Part Of ... 2000-09-02 semana2 87
10 2Ge+her The Hardest Part Of ... 2000-09-02 semana3 92
# ℹ 5,297 more rowsO número de linhas agora é muito menor, indicando que várias linhas que continham apenas NAs foram removidas.
Você também poderia estar se perguntando: o que aconteceria caso a música ficasse no top 100 por mais que 76 semanas? Não é possível dizer apenas com esses dados, mas você poderia imaginar que colunas adicionais semana77, semana78, … seriam incluídas no conjunto de dados.
Esses dados agora estão no formato tidy, mas nós ainda podemos facilitar cálculos futuros ao converter os valores da coluna semana de caracteres para números utilizando mutate() e readr::parse_number(). parse_number() é uma função bem útil que irá extrair o primeiro número de uma sequência de caracteres (string) e vai ignorar todo o resto.
top100musicas_longo <- top100musicas |>
pivot_longer(
cols = starts_with("semana"),
names_to = "semana",
values_to = "posicao",
values_drop_na = TRUE
) |>
mutate(
semana = parse_number(semana)
)
top100musicas_longo# A tibble: 5,307 × 5
artista musica data.adicionada semana posicao
<chr> <chr> <date> <dbl> <dbl>
1 2 Pac Baby Don't Cry (Keep... 2000-02-26 1 87
2 2 Pac Baby Don't Cry (Keep... 2000-02-26 2 82
3 2 Pac Baby Don't Cry (Keep... 2000-02-26 3 72
4 2 Pac Baby Don't Cry (Keep... 2000-02-26 4 77
5 2 Pac Baby Don't Cry (Keep... 2000-02-26 5 87
6 2 Pac Baby Don't Cry (Keep... 2000-02-26 6 94
7 2 Pac Baby Don't Cry (Keep... 2000-02-26 7 99
8 2Ge+her The Hardest Part Of ... 2000-09-02 1 91
9 2Ge+her The Hardest Part Of ... 2000-09-02 2 87
10 2Ge+her The Hardest Part Of ... 2000-09-02 3 92
# ℹ 5,297 more rowsAgora que temos todos os números das semanas em uma única variável e todas as posições em outra, é um bom ponto para visualizar como a posição das músicas varia ao longo do tempo. O código é mostrado abaixo e o resultado está em Figura 5.2. Nós podemos ver que muito poucas músicas permanecem no top 100 por mais de 20 semanas.
top100musicas_longo|>
ggplot(aes(x = semana, y = posicao, group = musica)) +
geom_line(alpha = 0.25) +
scale_y_reverse()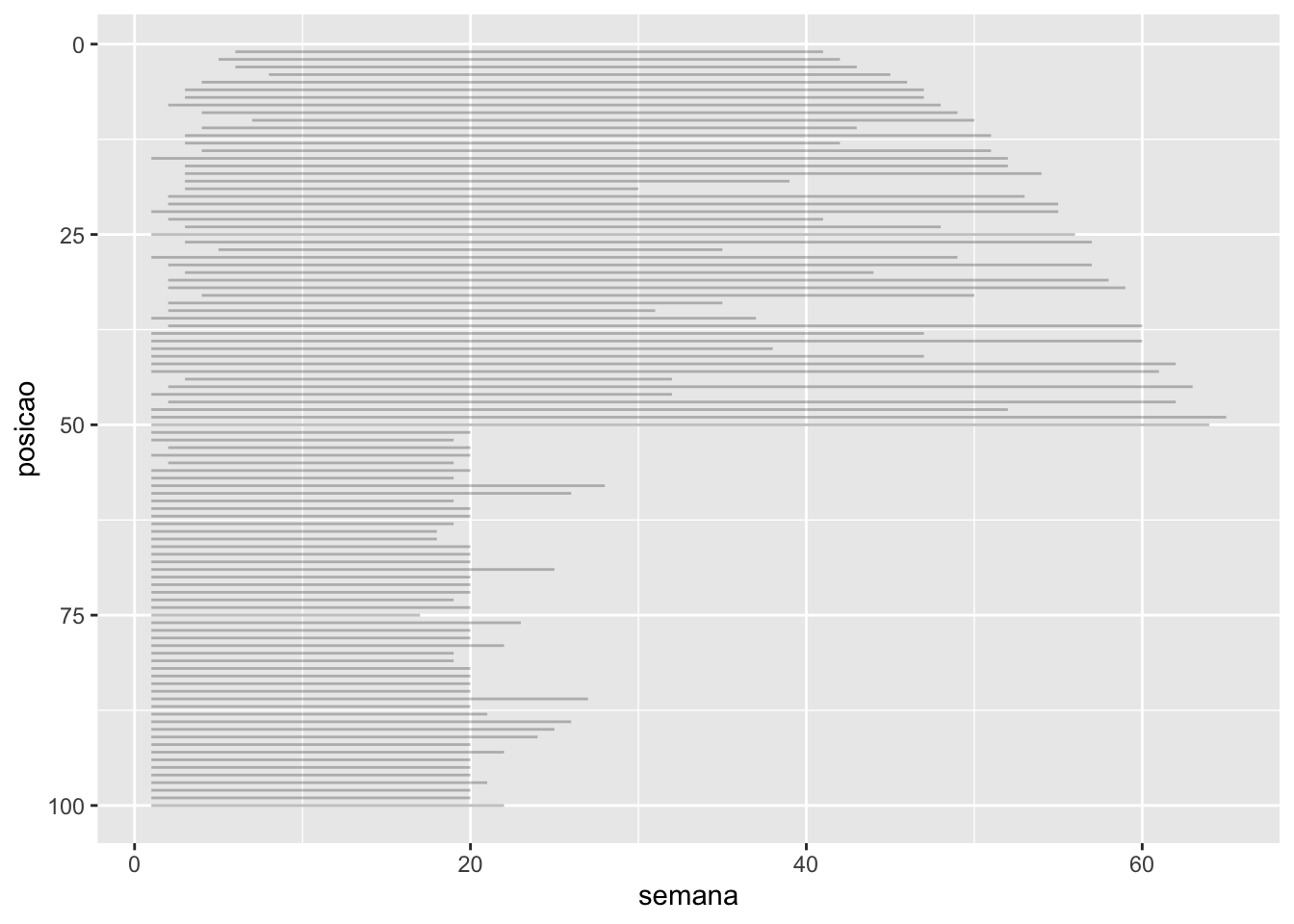
5.3.2 Como a pivotagem funciona?
Agora que você já viu como usar a pivotagem para mudar a forma de nossos dados, vamos tomar um tempinho para ganhar alguma intuição sobre o que a pivotagem faz com os dados. Vamos começar com um conjunto de dados bem simples para enxergamos com mais facilidade o que está acontecendo. Suponha que temos três pacientes com ids A, B e C, e fazemos a medição da pressão sanguínea (ps) duas vezes para cada paciente. Vamos criar os dados com tribble(), uma função útil para construir pequenos conjuntos de dados (tibbles) manualmente:
# ps - "pressão sanguínea"
df <- tribble(
~id, ~ps1, ~ps2,
"A", 100, 120,
"B", 140, 115,
"C", 120, 125
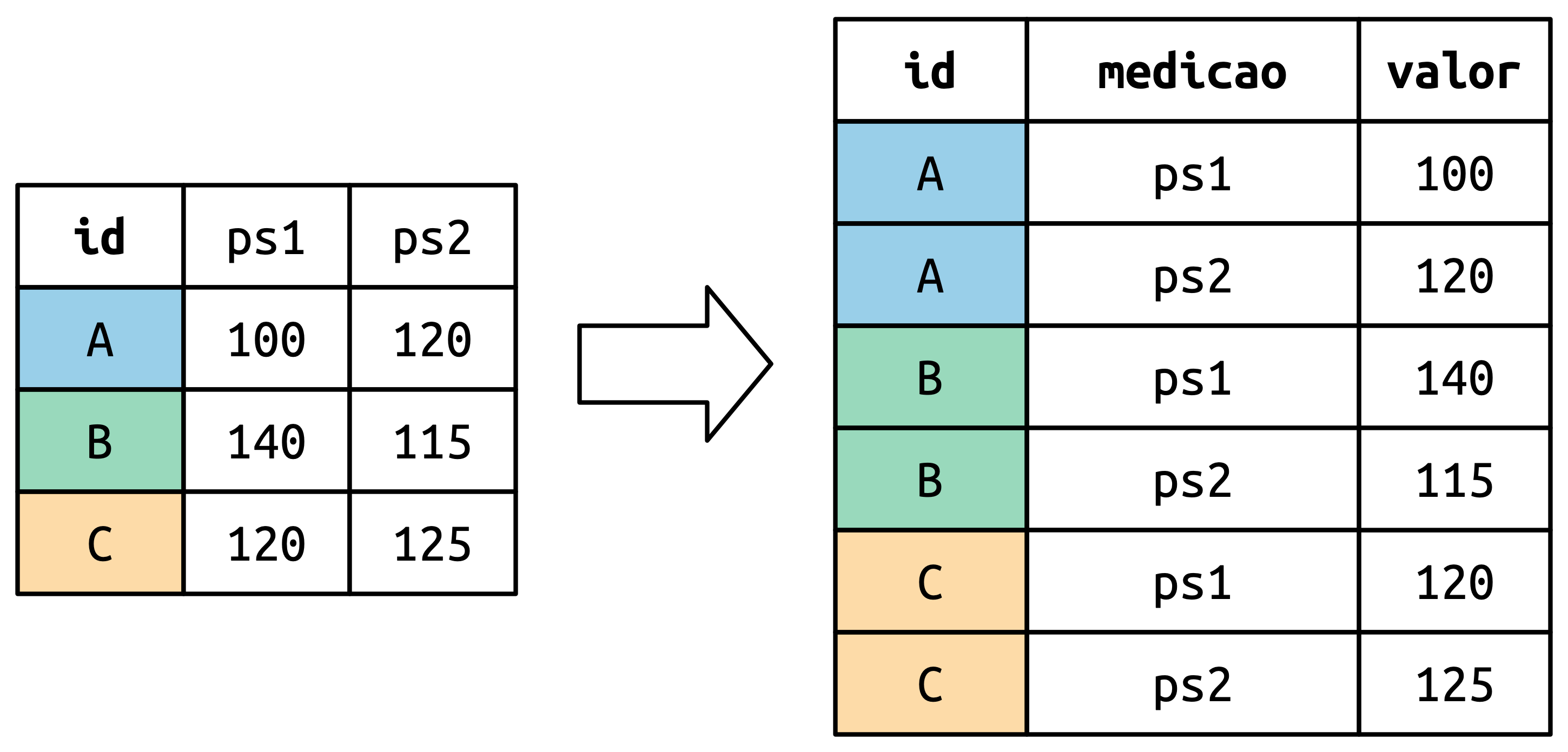
)Nós queremos que nosso conjunto de dados tenham três variáveis: id (já existe), medicao (o nome das colunas), e valor (valor das células). Para obter essa forma, precisamos pivotar df para um formato mais longo:
df |>
pivot_longer(
cols = ps1:ps2,
names_to = "medicao",
values_to = "valor"
)# A tibble: 6 × 3
id medicao valor
<chr> <chr> <dbl>
1 A ps1 100
2 A ps2 120
3 B ps1 140
4 B ps2 115
5 C ps1 120
6 C ps2 125Como funciona essa mudança de formato? É mais fácil de ver se pensarmos coluna por coluna. Como mostrado em Figura 5.3, os valores de uma coluna que já eram uma variável nos dados originais (id) precisam ser repetidos uma vez para cada coluna que foi pivotada.
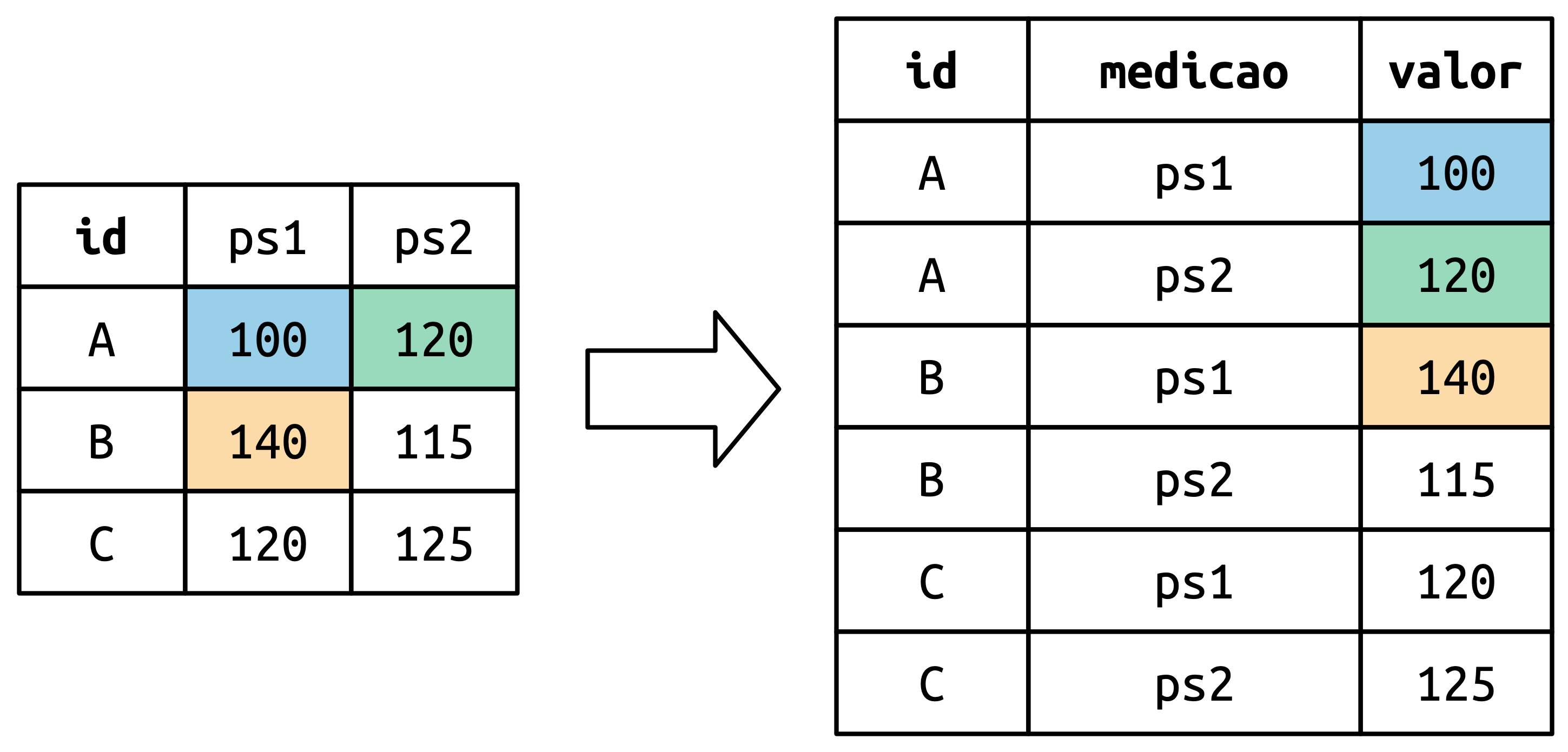
Os nomes das colunas se tornam valores em uma nova variável, cujo nome foi definido pelo parâmetro names_to, conforme mostrado em Figura 5.4. Eles precisam ser repetidos uma vez para cada linha dos dados originais.
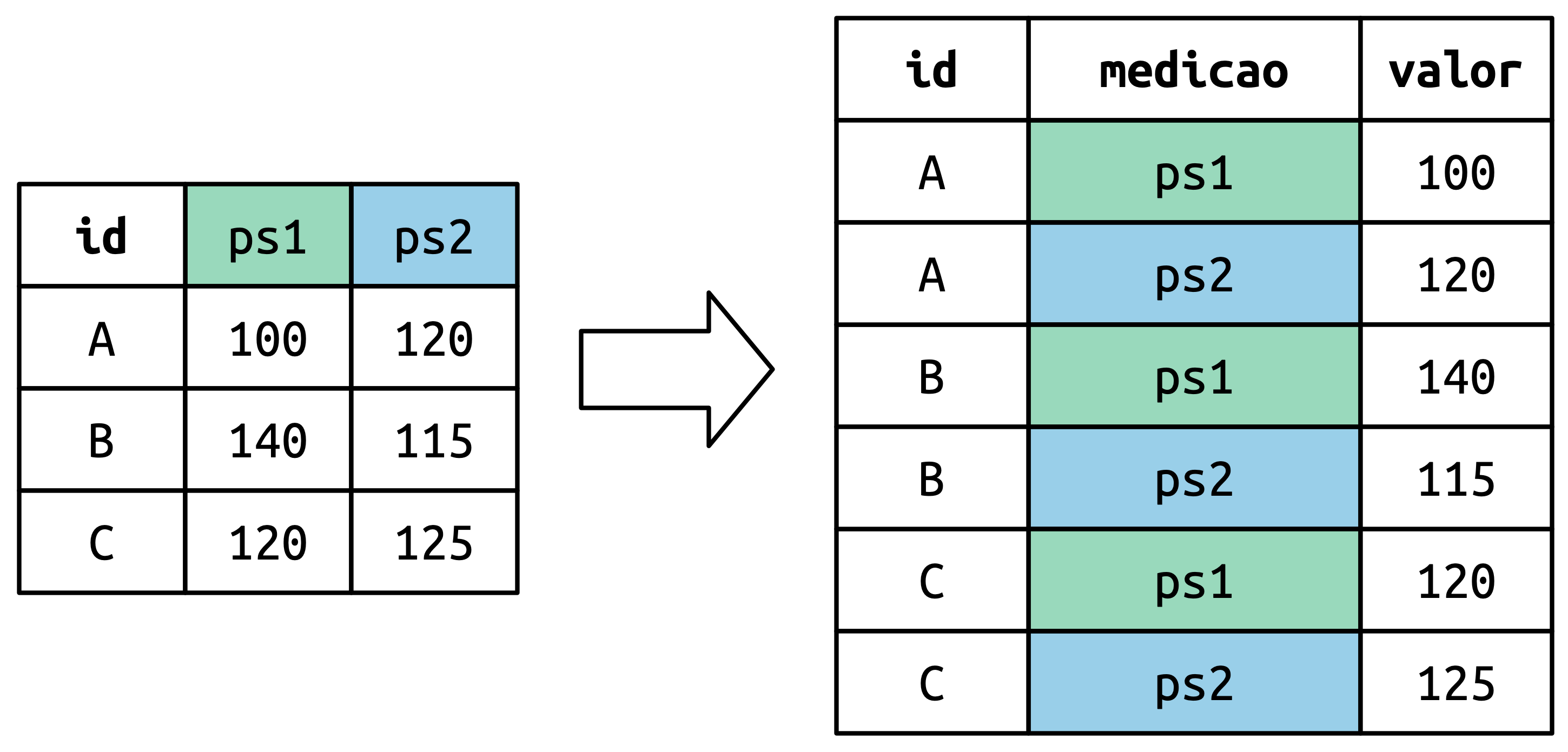
Os valores das células também se tornam valores em uma nova variável, com o nome definido por values_to. Eles são “desenrolados” linha a linha. Figura 5.5 ilustra o processo.
5.3.3 Muitas variáveis nos nomes de colunas
Uma situação mais desafiadora ocorre quando tem-se muitas partes de informação aglomeradas nos nomes das colunas, e você gostaria de armazená-las em variáveis separadas. Por exemplo, pegue o conjunto de dados dados_oms2, que é a fonte da tabela1 e outras similares que você viu acima:
dados_oms2# A tibble: 7,240 × 58
pais ano fpp_h_014 fpp_h_1524 fpp_h_2534 fpp_h_3544 fpp_h_4554 fpp_h_5564
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Afega… 1980 NA NA NA NA NA NA
2 Afega… 1981 NA NA NA NA NA NA
3 Afega… 1982 NA NA NA NA NA NA
4 Afega… 1983 NA NA NA NA NA NA
5 Afega… 1984 NA NA NA NA NA NA
6 Afega… 1985 NA NA NA NA NA NA
7 Afega… 1986 NA NA NA NA NA NA
8 Afega… 1987 NA NA NA NA NA NA
9 Afega… 1988 NA NA NA NA NA NA
10 Afega… 1989 NA NA NA NA NA NA
# ℹ 7,230 more rows
# ℹ 50 more variables: fpp_h_65 <dbl>, fpp_m_014 <dbl>, fpp_m_1524 <dbl>,
# fpp_m_2534 <dbl>, fpp_m_3544 <dbl>, fpp_m_4554 <dbl>, fpp_m_5564 <dbl>,
# fpp_m_65 <dbl>, fpn_h_014 <dbl>, fpn_h_1524 <dbl>, fpn_h_2534 <dbl>,
# fpn_h_3544 <dbl>, fpn_h_4554 <dbl>, fpn_h_5564 <dbl>, fpn_h_65 <dbl>,
# fpn_m_014 <dbl>, fpn_m_1524 <dbl>, fpn_m_2534 <dbl>, fpn_m_3544 <dbl>,
# fpn_m_4554 <dbl>, fpn_m_5564 <dbl>, fpn_m_65 <dbl>, ep_h_014 <dbl>, …Esse conjunto de dados, coletado pela Organização Mundial da Saúde (OMS)3, armazena informações sobre diagnósticos de tuberculose. Existem duas colunas que já são variáveis e parecem fáceis de interpretar: pais e ano. Em seguida vêm outras 56 colunas tais como fpp_m_014, fpn_m_4554 e ep_m_3544. Se você olhar com cuidado para essas colunas por tempo suficiente, verá que há um padrão. Cada nome de coluna é composto por três partes separadas por _. A primeira parte, sp/rel/ep, descreve o método utilizado para o diagnóstico, a segunda parte, h/m é o gênero - genero (codificado como uma variável binária nesse conjunto de dados), e a terceira parte, 014/1524/2534/3544/4554/5564/65 é o intervalo de idade - idade (014 representa 0-14, por exemplo).
Então, nesse caso temos seis partes de informação registrados em dados_oms2: o país e o ano (já nas colunas); o método diagnóstico, a categoria do gênero, e a categoria do intervalo de idade (contido nos outros nomes de colunas); e a contagem dos pacientes em naquela categoria (valores das células). Para organizar essas seis partes de informação em seis colunas separadas, utilizaremos pivot_longer() passando um vetor de nomes de colunas para o parâmetro names_to, instruções para separar, nas partes, os nomes originais das variáveis para o parâmetro names_sep e um nome de coluna para values_to:
dados_oms2 |>
pivot_longer(
cols = !(pais:ano),
names_to = c("diagnostico", "genero", "idade"),
names_sep = "_",
values_to = "contagem"
)# A tibble: 405,440 × 6
pais ano diagnostico genero idade contagem
<chr> <dbl> <chr> <chr> <chr> <dbl>
1 Afeganistão 1980 fpp h 014 NA
2 Afeganistão 1980 fpp h 1524 NA
3 Afeganistão 1980 fpp h 2534 NA
4 Afeganistão 1980 fpp h 3544 NA
5 Afeganistão 1980 fpp h 4554 NA
6 Afeganistão 1980 fpp h 5564 NA
7 Afeganistão 1980 fpp h 65 NA
8 Afeganistão 1980 fpp m 014 NA
9 Afeganistão 1980 fpp m 1524 NA
10 Afeganistão 1980 fpp m 2534 NA
# ℹ 405,430 more rowsUma alternativa a names_sep é names_pattern, que você pode usar para extrair variáveis em nomenclaturas mais complicadas, desde que você já tenha aprendido a usar expressões regulares Capítulo 15.
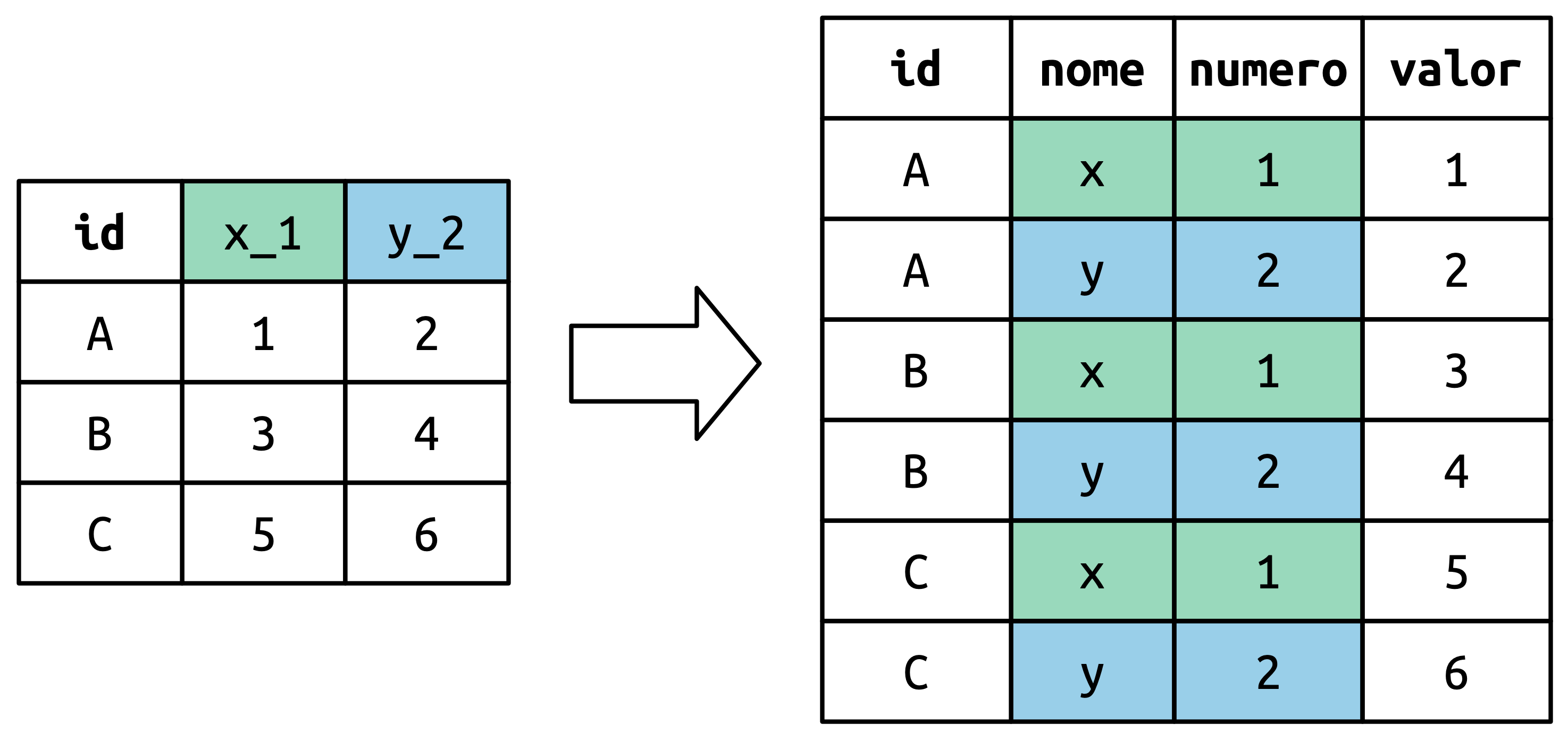
Conceitualmente, essa seria só uma pequena variação desse caso mais simples que você acabou de ver. Figura 5.6 mostra a ideia básica: agora, em vez da coluna de nomes pivotar para uma única coluna, ela pivota para múltiplas colunas. Você pode imaginar isso acontecendo em dois passos (primeiro pivotando e depois separando) mas no fundo, tudo ocorre em um único passo porque é mais rápido.
5.3.4 Dados e nomes de variáveis nos cabeçalhos das colunas
O próximo degrau em complexidade é quando os nomes das colunas incluem uma mistura de nomes e valores das variáveis. Por exemplo, vamos pegar o conjunto de dados nucleo_familiar:
nucleo_familiar# A tibble: 5 × 5
familia datanascimento_crianca1 datanascimento_crianca2 nome_crianca1
<int> <date> <date> <chr>
1 1 1998-11-26 2000-01-29 Susan
2 2 1996-06-22 NA Mark
3 3 2002-07-11 2004-04-05 Sam
4 4 2004-10-10 2009-08-27 Craig
5 5 2000-12-05 2005-02-28 Parker
# ℹ 1 more variable: nome_crianca2 <chr>Nesse conjunto temos dados de cinco famílias, com nomes e datas de nascimento de até duas crianças. O novo desafio é que nesses dados os nomes das colunas contêm os nomes de duas variáveis (data_nascimento, nome) e os valores de outra (criança - crianca, com valores 1 ou 2). Para resolver esse problema, novamente precisamos passar um vetor para names_to, mas dessa vamos vamos passar um parâmetro especial ".value" (chamado de sentinel); esse parâmetro não é o nome de uma variável, mas um valor único que diz ao pivot_longer() para fazer algo diferente com ele. Isso sobrepõe o argumento values_to normal em favor do primeiro componente do nome da coluna pivotada, utilizado como nome da variável na saída da função.
nucleo_familiar |>
pivot_longer(
cols = !familia,
names_to = c(".value", "crianca"),
names_sep = "_",
values_drop_na = TRUE
)# A tibble: 9 × 4
familia crianca datanascimento nome
<int> <chr> <date> <chr>
1 1 crianca1 1998-11-26 Susan
2 1 crianca2 2000-01-29 Jose
3 2 crianca1 1996-06-22 Mark
4 3 crianca1 2002-07-11 Sam
5 3 crianca2 2004-04-05 Seth
6 4 crianca1 2004-10-10 Craig
7 4 crianca2 2009-08-27 Khai
8 5 crianca1 2000-12-05 Parker
9 5 crianca2 2005-02-28 GracieNovamente utilizaremos values_drop_na = TRUE, uma vez que a forma da entrada força a criação de variáveis ausentes explícitas (ex: para famílias com apenas um filho).
Figura 5.7 ilustra a ideia básica em um exemplo mais simples. Quando você utiliza ".value" em names_to, a coluna de nomes dos dados de entrada contribui tanto para os valores quanto para os nomes das variáveis na saída.
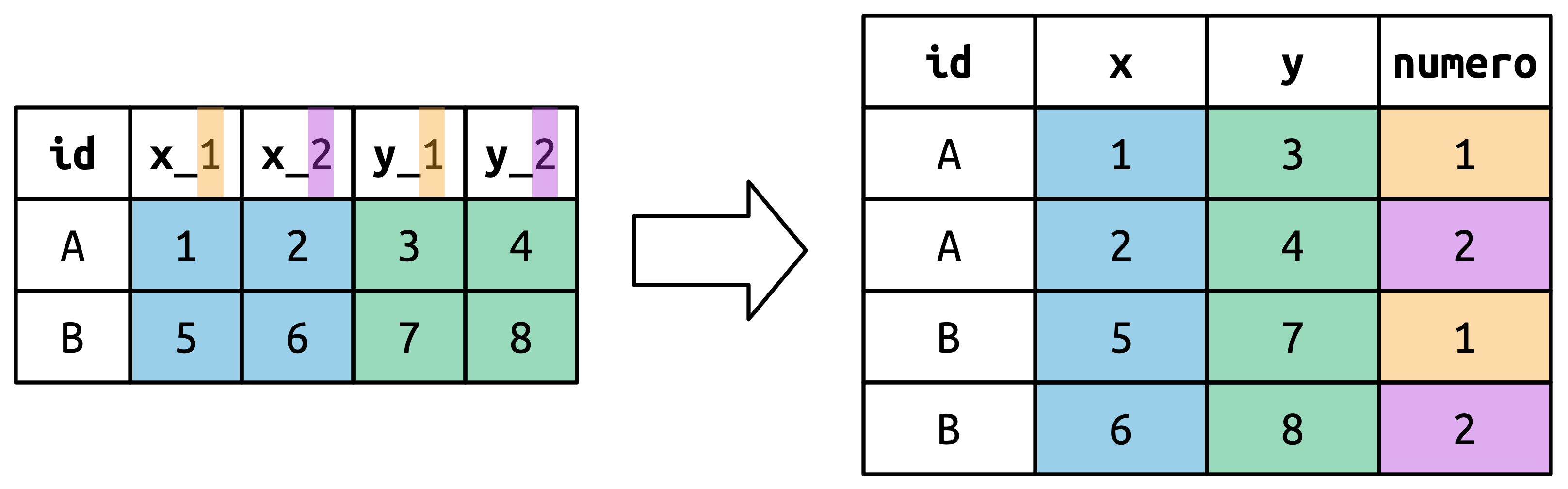
names_to = c(".value", "num") divide os nomes das colunas em dois componentes: a primeira parte determina o nome da coluna da saída, (x ou y), e a segunda parte determina o valore da coluna num.
5.4 Transformando os dados para o formato largo
Até então, utilizamos pivot_longer() para resolver um tipo comum de problemas no qual os valores foram movidos para os nomes de colunas. Agora vamos pivotar (HA HA) para pivot_wider(), que vai tornar o conjunto de dados mais largo (wider) ao aumentar o número de colunas e diminuir o número de linhas, auxiliando quando uma informação está espalhada em múltiplas linhas. Isso tende a aparecer menos na vida real, mas parece ser comum quando estamos lidando com dados governamentais.
Começaremos dando uma olhada em cms_paciente_experiencia, um conjunto de dados de serviços do Centers of Medicare and Medicaid que coleta dados sobre as experiências dos pacientes:
cms_paciente_experiencia# A tibble: 500 × 5
organizacao_id organizacao_nome medida_codigo medida_titulo taxa_performance
<chr> <chr> <chr> <chr> <dbl>
1 0446157747 USC CARE MEDICAL… CAHPS_GRP_1 Obtenção de … 63
2 0446157747 USC CARE MEDICAL… CAHPS_GRP_2 O quão bem o… 87
3 0446157747 USC CARE MEDICAL… CAHPS_GRP_3 CAHPS for MI… 86
4 0446157747 USC CARE MEDICAL… CAHPS_GRP_5 Promoção e e… 57
5 0446157747 USC CARE MEDICAL… CAHPS_GRP_8 Equipe do co… 85
6 0446157747 USC CARE MEDICAL… CAHPS_GRP_12 Administraçã… 24
7 0446162697 ASSOCIATION OF U… CAHPS_GRP_1 Obtenção de … 59
8 0446162697 ASSOCIATION OF U… CAHPS_GRP_2 O quão bem o… 85
9 0446162697 ASSOCIATION OF U… CAHPS_GRP_3 CAHPS for MI… 83
10 0446162697 ASSOCIATION OF U… CAHPS_GRP_5 Promoção e e… 63
# ℹ 490 more rowsA unidade principal sendo estudada é a organização, mas cada organização está espalhada em seis linhas, com uma linha para cada medição feita pela pesquisa. Nós podemos ver o conjunto completo dos valores de medida_codigo e medida_titulo utilizando a função distinct():
cms_paciente_experiencia |>
distinct(medida_codigo, medida_titulo)# A tibble: 6 × 2
medida_codigo medida_titulo
<chr> <chr>
1 CAHPS_GRP_1 Obtenção de cuidados, consultas e informações no momento certo
2 CAHPS_GRP_2 O quão bem os provedores se comunicam
3 CAHPS_GRP_3 CAHPS for MIPS SSM: Patient's Rating of Provider
4 CAHPS_GRP_5 Promoção e educação em saúde
5 CAHPS_GRP_8 Equipe do consultório cortês e prestativa
6 CAHPS_GRP_12 Administração dos recursos do paciente Nenhuma dessas colunas irá gerar nomes espetaculares: medida_codigo não dá nenhuma pista quanto ao significado da variável e medida_titulo é uma longa frase, com espaços, Por enquanto, nós utilizaremos medida_codigo como a fonte de nossa nova coluna de nomes, mas em uma análise real você deveria considerar seu próprio nome de variável, que deve ser curto e informativo.
pivot_wider() tem a interface oposta à pivot_longer(): em vez de escolher o novo nome das colunas, nós precisamos fornecer as colunas existentes que definem os valores de (values_from) e o nome da coluna (names_from):
cms_paciente_experiencia |>
pivot_wider(
names_from = medida_codigo,
values_from = taxa_performance
)# A tibble: 500 × 9
organizacao_id organizacao_nome medida_titulo CAHPS_GRP_1 CAHPS_GRP_2
<chr> <chr> <chr> <dbl> <dbl>
1 0446157747 USC CARE MEDICAL GROUP … Obtenção de … 63 NA
2 0446157747 USC CARE MEDICAL GROUP … O quão bem o… NA 87
3 0446157747 USC CARE MEDICAL GROUP … CAHPS for MI… NA NA
4 0446157747 USC CARE MEDICAL GROUP … Promoção e e… NA NA
5 0446157747 USC CARE MEDICAL GROUP … Equipe do co… NA NA
6 0446157747 USC CARE MEDICAL GROUP … Administraçã… NA NA
7 0446162697 ASSOCIATION OF UNIVERSI… Obtenção de … 59 NA
8 0446162697 ASSOCIATION OF UNIVERSI… O quão bem o… NA 85
9 0446162697 ASSOCIATION OF UNIVERSI… CAHPS for MI… NA NA
10 0446162697 ASSOCIATION OF UNIVERSI… Promoção e e… NA NA
# ℹ 490 more rows
# ℹ 4 more variables: CAHPS_GRP_3 <dbl>, CAHPS_GRP_5 <dbl>, CAHPS_GRP_8 <dbl>,
# CAHPS_GRP_12 <dbl>A saída ainda não parece muito correta; parece que ainda precisamos de múltiplas linhas para cada organização. Isso é por que também precisamos dizer à pivot_wider() qual a coluna ou colunas têm valores que identificam cada linha de forma única; nesse caso são as variáveis que começam com "org":
cms_paciente_experiencia |>
pivot_wider(
id_cols = starts_with("organizacao"),
names_from = medida_codigo,
values_from = taxa_performance
)# A tibble: 95 × 8
organizacao_id organizacao_nome CAHPS_GRP_1 CAHPS_GRP_2 CAHPS_GRP_3
<chr> <chr> <dbl> <dbl> <dbl>
1 0446157747 USC CARE MEDICAL GROUP INC 63 87 86
2 0446162697 ASSOCIATION OF UNIVERSITY… 59 85 83
3 0547164295 BEAVER MEDICAL GROUP PC 49 NA 75
4 0749333730 CAPE PHYSICIANS ASSOCIATE… 67 84 85
5 0840104360 ALLIANCE PHYSICIANS INC 66 87 87
6 0840109864 REX HOSPITAL INC 73 87 84
7 0840513552 SCL HEALTH MEDICAL GROUP … 58 83 76
8 0941545784 GRITMAN MEDICAL CENTER INC 46 86 81
9 1052612785 COMMUNITY MEDICAL GROUP L… 65 84 80
10 1254237779 OUR LADY OF LOURDES MEMOR… 61 NA NA
# ℹ 85 more rows
# ℹ 3 more variables: CAHPS_GRP_5 <dbl>, CAHPS_GRP_8 <dbl>, CAHPS_GRP_12 <dbl>Isso gera a saída que estávamos esperando.
5.4.1 Como funciona a função pivot_wider()?
Para entender como a função pivot_wider() funciona, vamos novamente começar com um conjunto de dados bem simples. Agora temos dois pacientes com ids A e B, e temos três medições de pressão sanguínea para o paciente A e duas para o paciente B:
df <- tribble(
~id, ~medida, ~valor,
"A", "ps1", 100,
"B", "ps1", 140,
"B", "ps2", 115,
"A", "ps2", 120,
"A", "ps3", 105
)Vamos pegar os valores da coluna valor e os nomes da coluna medida:
df |>
pivot_wider(
names_from = medida,
values_from = valor
)# A tibble: 2 × 4
id ps1 ps2 ps3
<chr> <dbl> <dbl> <dbl>
1 A 100 120 105
2 B 140 115 NAPara iniciar o processo, a função pivot_wider() precisa primeiramente entender o que será distribuído para as linhas e colunas. Os novos nomes de coluna serão os valores únicos de medida.
Por padrão, as linhas da saída são determinadas por todas as demais variáveis que não estão indo para os novos nomes ou valores. Essas são chamadas id_cols. Aqui temos apenas uma coluna, mas em geral, pode haver qualquer número delas.
A função pivot_wider() então combina esses resultados para gerar um data frame vazio:
# A tibble: 2 × 4
id x y z
<chr> <lgl> <lgl> <lgl>
1 A NA NA NA
2 B NA NA NA E em seguida, preenche todos os valores ausentes com os dados de entrada. Nesse caso, nem todas as células da saída possuem um valor correspondente na entrada visto que não temos a terceira medição de pressão para o paciente B, então aquela célula permanecerá vazia. Voltaremos a essa ideia de que a pivot_wider() pode “produzir” valores ausentes em Capítulo 18.
Você pode também estar se perguntando se há várias linhas na entrada que correspondem a apenas uma célula na saída. O exemplo abaixo, possui duas linhas que correspondem ao id “A” à medida “ps1”:
df <- tribble(
~id, ~medida, ~valor,
"A", "ps1", 100,
"A", "ps1", 102,
"A", "ps2", 120,
"B", "ps1", 140,
"B", "ps2", 115
)Se tentarmos pivotar essa tabela teremos uma saída que contém estruturas chamadas colunas de listas (list-columns), sobre as quais você aprenderá mais em Capítulo 23:
df |>
pivot_wider(
names_from = medida,
values_from = valor
)Warning: Values from `valor` are not uniquely identified; output will contain list-cols.
• Use `values_fn = list` to suppress this warning.
• Use `values_fn = {summary_fun}` to summarise duplicates.
• Use the following dplyr code to identify duplicates.
{data} |>
dplyr::summarise(n = dplyr::n(), .by = c(id, medida)) |>
dplyr::filter(n > 1L)# A tibble: 2 × 3
id ps1 ps2
<chr> <list> <list>
1 A <dbl [2]> <dbl [1]>
2 B <dbl [1]> <dbl [1]>Como você ainda não sabe como trabalhar com esse tipo de estrutura, você deve seguir as dicas que aparecem na mensagem de alerta (warning) para entender onde está o problema:
# A tibble: 1 × 3
id medida n
<chr> <chr> <int>
1 A ps1 2Aí então, você fica a cargo de entender o que deu de errado com os dados e ajustar os problemas já ocorridos ou utilizar suas habilidades de agrupar e resumir os dados originais para se certificar que que cada combinação dos valores de linhas e colunas estejam em apenas uma linha.
5.5 Resumo
Nesse capítulo você aprendeu sobre dados no formato tidy: dados que possuem variáveis nas colunas e observações nas linhas. Dados nesse formato facilitam o trabalho no tidyverse, pois sua estrutura consistente é entendida pela maioria das funções. O principal desafio, então, é transformar dados provenientes de quaisquer estruturas que você receber para o formato tidy. Para esse fim, você aprendeu sobre pivot_longer() e pivot_wider() que permitem a organização de vários conjuntos de dados que ainda não estejam no formato tidy. Os exemplos apresentados aqui são uma seleção dos vários presentes em vignette("pivot", package = "tidyr"), então, se você encontrar algum problema com o qual esse capítulo ainda não é capaz de te ajudar, é uma boa ideia checar nessa vignette4.
Um outro desafio é o fato de que para certos conjuntos de dados, pode ser impossível especificar se a versão longa (long) ou larga (wide) é a versão tidy. Isso é, em parte, um reflexo da nossa definição de dados tidy, em que dizemos que cada coluna deve conter uma variável, mas não definimos, de fato, o que é uma variável (e fazer isso é surpreendentemente difícil.) É totalmente aceitável dizer, de uma forma pragmática, que uma variável é qualquer coisa que facilite sua análise. Então, se tiver difícil descobrir como fazer algum tipo de cálculo ou análise, considere alterar a forma como seus dados estão organizados; não tenha medo de voltar para um formato untidy, transformá-lo e depois reorganizá-lo conforme necessário!
Se você gostou desse capítulo e quiser saber mais sobre a teoria por trás dele, você pode se aprofundar sobre a história e os fundamentos teóricos no artigo Tidy Data publicado no Journal of Statistical Software.
E agora que você já está escrevendo uma quantidade considerável de código em R, é a hora de aprender um pouco mais sobre como organizar o seu código em arquivos e diretórios. No próximo capítulo, você irá aprender sobre todas as vantagens dos scripts e projetos, e algumas das várias ferramentas que eles disponibilizam para tornar sua vida mais fácil.
A música será incluída aos dados desde que tenha estado no top 100 em algum momento do ano 2000, e então será monitorada por até 72 semanas após a primeira aparição.↩︎
Voltaremos a essas ideias em Capítulo 18.↩︎
Nota de tradução: do inglês World Health Organisation (WHO).↩︎
Nota de tradução: As vignettes são documentos que acompanham os pacotes do R e fornecem documentações mais extensas sobre o pacote e suas funções.↩︎