library(dados)
library(tidyverse)
#> ── Attaching core tidyverse packages ───────────────────── tidyverse 2.0.0 ──
#> ✔ dplyr 1.1.4.9000 ✔ readr 2.1.5
#> ✔ forcats 1.0.0 ✔ stringr 1.5.1
#> ✔ ggplot2 3.5.1 ✔ tibble 3.2.1
#> ✔ lubridate 1.9.4 ✔ tidyr 1.3.1
#> ✔ purrr 1.0.4
#> ── Conflicts ─────────────────────────────────────── tidyverse_conflicts() ──
#> ✖ dplyr::filter() masks stats::filter()
#> ✖ dplyr::lag() masks stats::lag()
#> ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors3 Transformação de dados
3.1 Introdução
Visualização de dados é uma ferramenta importante para gerar insights, mas é raro que você pegue os dados exatamente na forma que você precisa para fazer o gráfico que você quer. Geralmente você precisará criar algumas variáveis novas ou agregações para responder às perguntas com seus dados, ou talvez você precise apenas renomear as variáveis ou reordenar as observações para facilitar o trabalho com os dados. Você vai aprender a fazer tudo isso (e mais um pouco!) nesse capítulo, em que nós iremos lhe apresentar as possibilidades de transformações de dados utilizando o pacote dplyr e um novo conjunto de dados sobre vôos que partiram de Nova York em 2013.
O objetivo desse capítulo é te dar uma visão geral de todas as principais ferramentas para transformar um data frame. Começaremos com as funções que operam sobre as linhas e colunas de um data frame, em seguida voltaremos para falar mais um pouco sobre o pipe, que é uma importante ferramenta que você utilizará para combinar os verbos1 das transformações. Depois, iremos introduzir a habilidade de trabalhar com grupos. Finalizaremos o capítulo com um estudo de caso que demonstra todas essas funções em ação, e, mais tarde, voltaremos para vê-las em detalhe nos capítulos que seguem, quando começaremos a nos aprofundar em tipos mais específicos de dados (números, strings (de caracteres) e datas).
3.1.1 Pré-requisitos
Nesse capítulo focaremos no pacote dplyr, outro membro central do tidyverse. Ilustraremos as ideias principais utilizando dados do pacote dados, e visualizaremos com o ggplot2 para nos ajudar a entender os dados.
Preste bastante atenção às mensagens de conflito exibidas quando você carregar o tidyverse. Elas te indicam que o dplyr sobrescreve algumas funções do R base. Se você quiser utilizar a versão R base dessas funções depois de carregar o dplyr, você deverá usar seus nomes completos: stats::filter() e stats::lag(). Até agora nós basicamente ignoramos o pacote ao qual uma função pertence, pois na maioria das vezes isso não importava.
No entanto, conhecer o pacote pode te auxiliar na obtenção de ajuda e na busca por funções relacionadas, então quando for necessário sermos precisos a respeito de qual pacote uma função pertence, nós utilizaremos a mesma sintaxe que o R: nomedopacote::nomedafuncao().
3.1.2 Voos (nycflights13)
Para explorar os verbos básicos do dplyr iremos utilizar a base de dados dados::voos. Essa base contém todos os 336.776 vôos que saíram da cidade de Nova Iorque em 2013. Esses dados vêm de um órgão de transporte dos EUA chamado Bureau of Transportation Statistics, e estão documentados em ?dados::voos.
voos
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …voos é um tibble, um tipo especial de data frame utilizado pelo tidyverse para evitar algumas “pegadinhas” comuns. A diferença principal entre tibbles e data frames é forma como tibbles são exibidos no console; eles foram pensados para grandes conjuntos de dados, então eles mostram apenas algumas das primeiras linhas e somente as colunas que cabem na tela. Existem algumas opções para visualizar tudo. Se você estiver utilizando o RStudio, o mais conveniente provavelmente é View(voos), que vai abrir uma visualização interativa, com filtros e rolagem de tela. Caso não esteja, você pode utilizar print(voos, width = Inf) para mostrar todas as colunas, ou utilizar glimpse():
glimpse(voos)
#> Rows: 336,776
#> Columns: 19
#> $ ano <int> 2013, 2013, 2013, 2013, 2013, 2013, 2013, 2013, 20…
#> $ mes <int> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,…
#> $ dia <int> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,…
#> $ horario_saida <int> 517, 533, 542, 544, 554, 554, 555, 557, 557, 558, …
#> $ saida_programada <int> 515, 529, 540, 545, 600, 558, 600, 600, 600, 600, …
#> $ atraso_saida <dbl> 2, 4, 2, -1, -6, -4, -5, -3, -3, -2, -2, -2, -2, -…
#> $ horario_chegada <int> 830, 850, 923, 1004, 812, 740, 913, 709, 838, 753,…
#> $ chegada_prevista <int> 819, 830, 850, 1022, 837, 728, 854, 723, 846, 745,…
#> $ atraso_chegada <dbl> 11, 20, 33, -18, -25, 12, 19, -14, -8, 8, -2, -3, …
#> $ companhia_aerea <chr> "UA", "UA", "AA", "B6", "DL", "UA", "B6", "EV", "B…
#> $ voo <int> 1545, 1714, 1141, 725, 461, 1696, 507, 5708, 79, 3…
#> $ codigo_cauda <chr> "N14228", "N24211", "N619AA", "N804JB", "N668DN", …
#> $ origem <chr> "EWR", "LGA", "JFK", "JFK", "LGA", "EWR", "EWR", "…
#> $ destino <chr> "IAH", "IAH", "MIA", "BQN", "ATL", "ORD", "FLL", "…
#> $ tempo_voo <dbl> 227, 227, 160, 183, 116, 150, 158, 53, 140, 138, 1…
#> $ distancia <dbl> 1400, 1416, 1089, 1576, 762, 719, 1065, 229, 944, …
#> $ hora <dbl> 5, 5, 5, 5, 6, 5, 6, 6, 6, 6, 6, 6, 6, 6, 6, 5, 6,…
#> $ minuto <dbl> 15, 29, 40, 45, 0, 58, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
#> $ data_hora <dttm> 2013-01-01 05:00:00, 2013-01-01 05:00:00, 2013-01…Em ambas as visualizações, o nome das variáveis estão seguidos por abreviaturas que te indicam o tipo de cada variável: <int> para inteiros, <dbl> double2 (números reais), <chr> para caracteres (strings) e <dttm> para date-time (data e hora). Isso é importante pois as operações possíveis sobre uma coluna depende muito do seu “tipo”.
3.1.3 Básico do dplyr
Você está prestes a aprender os verbos (funções) primários do dplyr que já vão lhe permitir resolver a vasta maioria dos desafios de manipulação de dados. Mas antes de discutirir suas diferenças individuais, é importante destacar o que eles têm em comum:
O primeiro argumento é sempre um data frame.
Os demais argumentos geralmente descrevem sobre quais colunas a operação será executada, utilizando o nome das variáveis (sem aspas).
A saída é sempre um novo data frame.
Pelo fato de cada verbo fazer bem apenas UMA coisa, a resolução de problemas complexos normalmente requer a combinação de múltiplos verbos. Fazemos isso utilizando o pipe: |>.
Falaremos mais do pipe em Seção 3.4, mas resumindo, o pipe recebe alguma coisa à sua esquerda e repassa essa coisa para a função à sua direita, de forma que x |> f(y) seja equivalente a f(x, y), e x |> f(y) |> g(z) seja equivalente a g(f(x, y), z). A forma mais fácil de pronunciar o pipe é “então” (“then”). Isso torna possível pegar a visão geral do código abaixo, mesmo que você ainda não tenha aprendido os detalhes:
Verbos do dplyr são organizados em quatro grupos baseados na estrutura sobre a qual operam: linhas (rows), colunas (columns), grupos (groups) ou tabelas (tables). Nas próximas seções você aparenderá os verbos mais importantes para linhas, colunas e grupos, então voltaremos para os verbos de join que operam em tabelas na seção Capítulo 19. Vamos nessa!
3.2 Linhas (rows)
Os verbos mais importantes que operam nas linhas de um conjunto de dados são filter(), que modifica quais linhas estão presentes sem mudar a sua ordem, e arrange(), que modifica a ordem das linhas sem modificar quais estão presentes. Ambas as funções afetam apenas as linhas e as colunas permanecem intocadas. Também vamos falar sobre distinct(), que acha as linhas com valores únicos, mas diferentemente de arrange() e filter() também pode, opcionalmente, modificar as colunas.
3.2.1 filter()
filter() lhe permite manter linhas com base nos valores das colunas3. O primeiro argumento é o data frame. O segundo e demais argumentos são as condições que devem ser verdadeiras para manter a linha. Por exemplo, podemos achar todos os vôos que decolaram com mais de 120 minutos (duas horas) de atraso:
voos |>
filter(atraso_saida > 120)
#> # A tibble: 9,723 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 848 1835 853
#> 2 2013 1 1 957 733 144
#> 3 2013 1 1 1114 900 134
#> 4 2013 1 1 1540 1338 122
#> 5 2013 1 1 1815 1325 290
#> 6 2013 1 1 1842 1422 260
#> # ℹ 9,717 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Assim como > (maior que), você também pode usar >= (maior ou igual a), < (menor que), <= (menor ou igual a), == (igual a), e != (diferente de). Você também pode combinar condições com & ou , para indicar “e” (avalia ambas as condições) ou com | para indicar “ou” (avalia ao menos uma condição):
# Voos que partiram no dia 1 de Janeiro
voos |>
filter(mes == 1 & dia == 1)
#> # A tibble: 842 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 836 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …
# Voos que partiram em janeiro ou fevereiro
voos |>
filter(mes == 1 | mes == 2)
#> # A tibble: 51,955 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 51,949 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Existe um atalho útil quando você está combinando | e ==: %in%. Ele vai manter as linhas cujas variáveis são iguais a um dos valores à direita:
# Uma forma mais curta de selecionar vôos que decolaram em janeiro ou fevereiro
voos |>
filter(mes %in% c(1, 2))
#> # A tibble: 51,955 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 51,949 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Vamos voltar para essas comparações e operadores lógicos com mais detalhes em Capítulo 12.
Quando você roda filter() o dplyr executa a operação de filtragem, criando um novo data frame, e em seguida o exibe. Ele não modifica o conjunto de dados voos já existente, pois as funções do dplyr nunca modificam seus argumentos de entrada. Para salvar o resultado, você precisa utilizar o operador de atribuição <-:
jan1 <- voos |>
filter(mes == 1 & dia == 1)3.2.2 Erros comuns
Quando você está começando com o R, o erro mais fácil de se cometer é utilizar = em vez de == quando se está testando igualdade. filter() vai te avisar quando isso acontecer:
voos |>
filter(mes = 1)
#> Error in `filter()`:
#> ! We detected a named input.
#> ℹ This usually means that you've used `=` instead of `==`.
#> ℹ Did you mean `mes == 1`?Outro erro é escrever as expressões com “ou” como faríamos em Português:
voos |>
filter(mes == 1 | 2)Isso até “funciona”, no sentido de que não retornará um erro, mas ele não vai fazer o que você esperaria, pois | primeiro vai avaliar a condição mes == 1 e depois a condição 2, que não é uma condição adequada para se avaliar nesse caso. Iremos aprender mais sobre o que está acontecendo aqui em Seção 15.6.2.
3.2.3 arrange()
arrange() modifica a ordem das linhas com base no valor das colunas. Ela recebe um data frame e um conjunto de nomes de colunas (ou expressões mais complicadas) para ordenar por eles. Se você fornecer mais de um nome de coluna, cada coluna a mais será utilizada para desempatar os valores das colunas anteriores. Por exemplo, o código a seguir vai ordenar por horário da decolagem, que está distribuído em 4 colunas. Nós pegamos primeiro os anos mais antigos e então, dentro de cada ano, os primeiros meses e assim por diante.
voos |>
arrange(ano, mes, dia, horario_saida)
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Você pode utilizar desc() em uma coluna dentro de arrange() para re-ordenar o data frame com base naquele coluna em ordem decrescente (do maior para o menor). Por exemplo, esse código ordena os vôos do mais para os menos atrasados:
voos |>
arrange(desc(atraso_saida))
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 9 641 900 1301
#> 2 2013 6 15 1432 1935 1137
#> 3 2013 1 10 1121 1635 1126
#> 4 2013 9 20 1139 1845 1014
#> 5 2013 7 22 845 1600 1005
#> 6 2013 4 10 1100 1900 960
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Perceba que o número de linhas não se alterou – estamos apenas organizando os dados e não filtrando.
3.2.4 distinct()
distinct() acha todas as linhas únicas em um conjunto de dados, então, do ponto de vista técnico ela opera principialmente nas linhas. No entanto, na maioria das vezes você vai querer combinações distintas de algumas variáveis, então você pode, opcionalmente, fornecer nome de colunas:
# Remove linhas duplicadas, se existirem
voos |>
distinct()
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …
# Acha todos os pares únicos de origens e destinos
voos |>
distinct(origem, destino)
#> # A tibble: 224 × 2
#> origem destino
#> <chr> <chr>
#> 1 EWR IAH
#> 2 LGA IAH
#> 3 JFK MIA
#> 4 JFK BQN
#> 5 LGA ATL
#> 6 EWR ORD
#> # ℹ 218 more rowsPor outro lado, caso você queira manter outras colunas quando estiver filtrando para linhas únicas, você pode usar a opção .keep_all = TRUE.
voos |>
distinct(origem, destino, .keep_all = TRUE)
#> # A tibble: 224 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 218 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Não é uma coincidência que todos esses vôos distintos sejam em 1º de janeiro: distinct() irá achar a primeira ocorrência de uma linha única no conjunto de dados e decartar todas as demais.
Se, em vez disso, você quiser achar o número de ocorrências, é melhor trocar distinct() por count(), e com o argumento sort = TRUE você pode organizá-los em ordem decrescente do número de ocorrências. Você vai aprendar mais sobre contagens em Seção 13.3.
voos |>
count(origem, destino, sort = TRUE)
#> # A tibble: 224 × 3
#> origem destino n
#> <chr> <chr> <int>
#> 1 JFK LAX 11262
#> 2 LGA ATL 10263
#> 3 LGA ORD 8857
#> 4 JFK SFO 8204
#> 5 LGA CLT 6168
#> 6 EWR ORD 6100
#> # ℹ 218 more rows3.2.5 Exercícios
-
Utilizando um único pipeline para cada condição, ache todos os vôos que cumpram cada condição:
- Teve um atraso na chegada de duas ou mais horas
- Voou para Houston (
IAHouHOU) - Foi operado pela United, American ou Delta
- Decolou no verão4 (julho, agosto e setembro)
- Chegou com mais de duas horas de atraso, mas não teve atraso na decolagem.
- Atrasou pelo menos uma hora, mas recuperou mais de 30 minutos em vôo.
Ordene
voospara achar os vôos com os maiores atrasos na decolagem. Ache os vôos que saíram o mais cedo pela manhã.Ordene
voospara achar os vôos mais rápidos. (Dica: tente incluir algum cálculo matemático dentro da sua função.)Houve pelo menos um vôo em cada dia de 2013?
Quais vôos percorream as maiores distâncias? Quais percorreram as menores?
Faz diferença a ordem em que você usa
filter()earrange()se você estiver utilizando ambos? Por que/por que não? Reflita sobre os resultados e quanto trabalho as funções teriam que executar.
3.3 Colunas (columns)
Existem quatro verbos importantes que afetam as colunas sem modificar as linhas: mutate() cria novas colunas que são derivadas das colunas existentes, select() alteram as colunas que estão presentes, rename() modifica o nome das colunas e relocate() modifica as posições das colunas.
3.3.1 mutate()
O papel de mutate() é adicionar novas colunas que são calculadas a partir de colunas existentes. Nos capítulos sobre transformações, você aprenderá sobre um grande conjunto de funções que podem ser utilizadas para manipular diferentes tipos de variáveis. Por enquanto, vamos nos ater à álgebra básica, que nos permite calcular o tempo_ganho, ou seja, quanto tempo um vôo atrasado recuperou no ar, e a velocidade velocidade em milhas por hora:
voos |>
mutate(
tempo_ganho = atraso_saida - atraso_chegada,
velocidade = distancia / tempo_voo * 60
)
#> # A tibble: 336,776 × 21
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 15 more variables: horario_chegada <int>, chegada_prevista <int>, …Por padrão, mutate() adiciona novas colunas à direita do seu conjunto de dados, o que torna difícil ver o que está acontecendo aqui. Podemos usar o argumento .before para, em vez disso, adicionar as variáveis à esquerda5:
voos |>
mutate(
tempo_ganho = atraso_saida - atraso_chegada,
velocidade = distancia / tempo_voo * 60,
.before = 1
)
#> # A tibble: 336,776 × 21
#> tempo_ganho velocidade ano mes dia horario_saida saida_programada
#> <dbl> <dbl> <int> <int> <int> <int> <int>
#> 1 -9 370. 2013 1 1 517 515
#> 2 -16 374. 2013 1 1 533 529
#> 3 -31 408. 2013 1 1 542 540
#> 4 17 517. 2013 1 1 544 545
#> 5 19 394. 2013 1 1 554 600
#> 6 -16 288. 2013 1 1 554 558
#> # ℹ 336,770 more rows
#> # ℹ 14 more variables: atraso_saida <dbl>, horario_chegada <int>, …O . é um sinal que mostra que .before é um argumento da função e não o nome de uma terceira variável que estamos criando. Você também pode utilizar .after para adicionar após uma variável, e tanto com .before quanto com .after você pode utilizar o nome da variável em vez da posição. Por exemplo, nós podemos adicionar novas variáveis após dia:
voos |>
mutate(
tempo_ganho = atraso_saida - atraso_chegada,
velocidade = distancia / tempo_voo * 60,
.after = dia
)Ou então, você pode controlar quais variáveis são mantidas com o argumento .keep. Um argumento particularmente útil é "used" que especifica que nós queremos manter apenas as colunas que estiveram envolvidas ou foram criadas pela etapa de mutate(). Por exemplo, a saída a seguir conterá apenas as variáveis atraso_saida, atraso_chegada, tempo_voo, tempo_ganho, horas e tempo_ganho_per_hour.
voos |>
mutate(
tempo_ganho = atraso_saida - atraso_chegada,
horas = tempo_voo / 60,
tempo_ganho_por_hora = tempo_ganho / horas,
.keep = "used"
)Perceba que, uma vez que nós não atribuímos o resultado da operação de volta a voos, as novas variáveis tempo_ganho, horas e tempo_ganho_por_hora serão apenas exibidas no console mas não serão armazenadas em um data frame. Se nós quisermos tê-las disponíveis em um data frame para usá-las no futuro, devemos pensar com cuidado a respeito de atribuir o resultado de volta a voos, sobrescrevendo o data frame original com muito mais variáveis, ou em um novo objeto. Normalmente, a resposta correta é utilizar um novo objeto, que será nomeado de forma informativa para indicar seu conteúdo, ex: ganho_atraso, mas você talvez tenha boas razões para sobrescrever voos.
3.3.2 select()
Não é incomum obter conjuntos de dados com centenas ou até mesmo milhares de variáveis. Nessa situação, o primeiro desafio tende as ser focar apenas nas variáveis que você está interessado. select() permite que você rapidamente dê um zoom (foque) em um subconjunto adequado utilizando operações baseadas no nome das variáveis:
-
Seleciona colunas por nome:
voos |> select(ano, mes, dia) -
Seleciona todas as colunas entre ano e dia (inclusos):
voos |> select(ano:dia) -
Seleciona todas as colunas exceto aquelas entre ano e dia (inclusos):
voos |> select(!ano:dia)
Historicamente, essa operação era feita com - em vez de !, então é provável que você veja isso por aí. Esses dois operadores servem ao mesmo propósito, mas com diferenças sutis no comportamento. Recomendamos usar ! porque é lido como “não” e se combina bem com & e |.
-
Seleciona todas as colunas que são caracteres:
Existe uma variedade de funções auxiliares que você pode utilizar junto com select():
-
starts_with("abc"): dá match em nomes que se iniciam com “abc”. -
ends_with("xyz"): dá match em nomes que terminam “xyz”. -
contains("ijk"): dá match em nomes que contém “ijk”. -
num_range("x", 1:3): buscaráx1,x2ex3.
Confira ?select para mais detalhes. Uma vez que você saiba expressões regulares (o tópico de Capítulo 15) você também poderá utilizar matches() para selecionar variáveis que são identificáveis por um dado padrão.
Você pode renomear variáveis à media que seleciona elas com select() utilizando =. O novo nome aparece à esquerda do = e o nome antigo à direita:
voos |>
select(tail_num = codigo_cauda)
#> # A tibble: 336,776 × 1
#> tail_num
#> <chr>
#> 1 N14228
#> 2 N24211
#> 3 N619AA
#> 4 N804JB
#> 5 N668DN
#> 6 N39463
#> # ℹ 336,770 more rows
3.3.3 rename()
Se você quiser manter todas as variáveis disponíveis e quer renomear apenas algumas delas, você pode utilizar rename() em vez de select():
voos |>
rename(tail_num = codigo_cauda)
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Se você tem várias colunas com nomes inconsistentes e gostaria de manualmente consertá-las de uma vez, confira o pacote janitor::clean_names() que fornece alguns métodos úteis de limpeza.
3.3.4 relocate()
Utilize relocate() para mover as variáveis de lugar. Talvez você queira deixar variáveis relacionadas juntas ou mover variáveis importantes para o início do data frame. Por padrão relocate() já move as variáveis para o início:
voos |>
relocate(data_hora, tempo_voo)
#> # A tibble: 336,776 × 19
#> data_hora tempo_voo ano mes dia horario_saida
#> <dttm> <dbl> <int> <int> <int> <int>
#> 1 2013-01-01 05:00:00 227 2013 1 1 517
#> 2 2013-01-01 05:00:00 227 2013 1 1 533
#> 3 2013-01-01 05:00:00 160 2013 1 1 542
#> 4 2013-01-01 05:00:00 183 2013 1 1 544
#> 5 2013-01-01 06:00:00 116 2013 1 1 554
#> 6 2013-01-01 05:00:00 150 2013 1 1 554
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: saida_programada <int>, atraso_saida <dbl>, …Você também pode especificar onde quer colocá-las utilizando .before e .after, assim como em mutate():
voos |>
relocate(ano:horario_saida, .after = data_hora)
voos |>
relocate(starts_with("cheg"), .before = horario_saida)3.3.5 Exercícios
Compare
horario_saida,saida_programada, eatraso_saida. Como você esperaria que esses três números estivessem relacionados?Esboce a maior quantidade possível de maneiras de selecionar
horario_saida,atraso_saida,horario_chegada, eatraso_chegadaa partir devoos.O que acontece se você especificar o nome de uma mesma variável múltiplas vezes em uma chamada à
select()?-
O que a função
any_of()faz? Por que ela pode ser útil se utilizada em conjunto com esse vetor?variables <- c("ano", "mes", "dia", "atraso_saida", "atraso_chegada") -
O resultado de rodar o código a seguir te surpreende de alguma forma? Como que que as funções auxiliares de seleção lidam, por padrão, com maiúsculas e minúsculas? Como você pode alterar esse padrão?
Renomeie
tempo_vooparatempo_voo_minpara indicar as unidades de medida e mova-a para o início do data frame.-
Por que o código abaixo não funciona e o que o erro significa?
3.4 O pipe
Nós te mostramos acima alguns exemplos simples do uso do pipe, mas o seu real poder emerge quando você começa a combinar múltiplos verbos. Por exemplo, imagine que você gostaria de achar os vôos mais rápidos do aeroporto IAH em Houston: você precisaria combinar filter(), mutate(), select() e arrange():
voos |>
filter(destino == "IAH") |>
mutate(velocidade = distancia / tempo_voo * 60) |>
select(ano:dia, horario_saida, companhia_aerea, voo, velocidade) |>
arrange(desc(velocidade))
#> # A tibble: 7,198 × 7
#> ano mes dia horario_saida companhia_aerea voo velocidade
#> <int> <int> <int> <int> <chr> <int> <dbl>
#> 1 2013 7 9 707 UA 226 522.
#> 2 2013 8 27 1850 UA 1128 521.
#> 3 2013 8 28 902 UA 1711 519.
#> 4 2013 8 28 2122 UA 1022 519.
#> 5 2013 6 11 1628 UA 1178 515.
#> 6 2013 8 27 1017 UA 333 515.
#> # ℹ 7,192 more rowsMesmo que esse pipeline tenha quatro etapas, é fácil passar o olho e entendê-lo pois os verbos vêm no início de cada linha: começa com os dados de voos, então filtra (filter), então modifica (mutate), então seleciona (select), então ordena(arrange).
O que aconteceria se não tivéssemos o pipe? Poderíamos aninhar cada chamada de função dentro da chamada anterior:
Ou poderíamos utilizar alguns objetos intermediários:
Ainda que ambas as formas tenham sua hora e lugar, o pipe geralmente produz o código de análise de dados que é mais fácil de escrever e ler.
Para adicionar o pipe ao seu código, recomendamos utilizar o atalho nativo do teclado: Ctrl/Cmd + Shift + M. Você terá que fazer uma mudança nas opções do RStudio para utilizar |> em vez de %>% conforme mostrado em Figura 3.1; mais sobre %>% em breve.
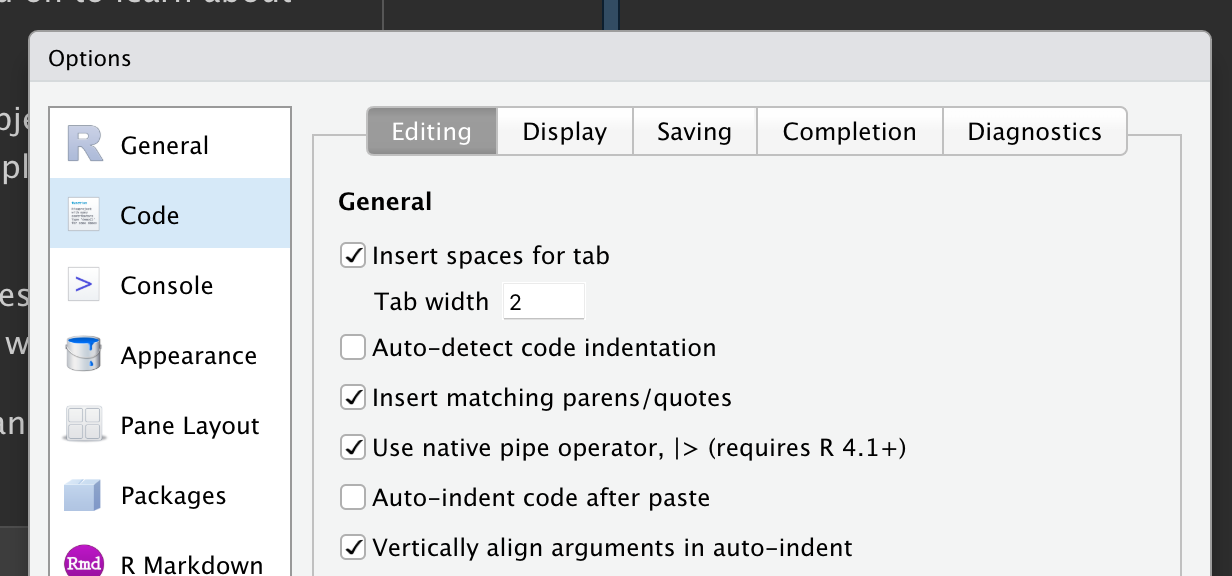
|>, certifique-se de que a opção “Use native pipe operator” está marcada.
Se você vem utilizando o tidyverse há um tempo, você deve estar familiarizado com o pipe %>% fornecido pelo pacote magrittr. O pacote magrittr está incluído nos pacotes principais do tidyverse, então você pode utilizar %>% sempre que carregar o tidyverse:
Para casos simples, |> e %>% se comportam de forma idêntica. Então por que recomendamos o pipe do R base? Primeiramente, porque o fato de ser parte do R base, significa que ele está sempre disponível para uso, mesmo que você não esteja utilizando o tidyverse. Em segundo lugar, |> é bem mais simples que %>%: nesse meio tempo entre a invenção do %>% em 2014 e a inclusão do |> no R 4.1.0 em 2021, aprendemos muita coisa sobre o pipe. Isso permitiu que a implementação no R base se livrasse de funcionalidades com usos pouco frequentes e menos importantes.
3.5 Grupos (groups)
Até aqui você aprendeu sobre funções que trabalham com linhas e colunas. dplyr se torna ainda mais poderoso quando você acrescenta a habilidade de trabalhar com grupos. Nessa seção, focaremos nas funções mais importantes: group_by(), summarize(), e a família de funções slice.
3.5.1 group_by()
Utilize group_by() para dividir o seu conjunto de dados em grupos que tenham algum significado para sua análise:
voos |>
group_by(mes)
#> # A tibble: 336,776 × 19
#> # Groups: mes [12]
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …group_by() não modifica seus dados mas, se você observar a saída de perto, você verá que a há uma indicação de que ela está agrupada (“grouped by”) mês (Groups: mes [12]). Isso significa que todas as operações subsequentes irão operar “por mês”. group_by() adiciona essa funcionalidade agrupada (“grouped”) (referenciada como uma classe) ao data frame, que modifica o comportamento dos verbos subsequentes que são aplicados aos dados.
3.5.2 summarize()
A operação agrupada mais importante é a sumarização (“summary”), que caso esteja sendo utilizada para calcular uma única sumarização estatística, reduz o data frame para ter apenas uma única linha para cada grupo.
No dplyr, essa operação é feita pela função summarize()6, como mostrado pelo exemplo a seguir, que calcula o atraso médio das decolagens por mês:
Uow! Alguma coisa deu errado e todos os nosso resultados viraram NAs (pronuncia-se “N-A”), que é o símbolo do R para valores ausentes. Isso ocorreu pois alguns dos vôos observados possuíam dados ausentes na coluna de atrasos, e por isso, quando calculamos a média incluindo esses valores, obtemos um NA como resultado. Voltaremos a falar de valores ausentes em detalhes em Capítulo 18, mas por enquanto, vamos pedir à função mean() para ignorar todos os valores ausentes definindo o argumento na.rm como TRUE:
Você pode criar quantas sínteses quiser em uma única chamada à summarize(). Você irá aprender várias sumarizações úteis nos próximos capítulos, mas uma que é muito útil é n(), que retorna o número de linhas de cada grupo.
Surpreendentemente, médias e contagens podem te levar longe em ciência de dados!
3.5.3 As funções slice_
Existem cinco funções úteis que lhe permite extrair linhas específicas de dentro de cada grupo:
-
df |> slice_head(n = 1)pega a primeira linha de cada grupo. -
df |> slice_tail(n = 1)pega a última linha de cada grupo. -
df |> slice_min(x, n = 1)pega a linha com o menor valor da colunax. -
df |> slice_max(x, n = 1)pega a linha com o maior valor da colunax. -
df |> slice_sample(n = 1)pega uma linha aleatória.
Você pode variar n para selecionar mais do que uma linha, ou, em vez de usar n =, você pode usar prop = 0.1 para selecionar (por exemplo) 10% das linhas de cada grupo. Por exemplo, o código a seguir acha os vôos que estão mais atrasados na chegada em cada destino.
voos |>
group_by(destino) |>
slice_max(atraso_chegada, n = 1) |>
relocate(destino)
#> # A tibble: 108 × 19
#> # Groups: destino [105]
#> destino ano mes dia horario_saida saida_programada atraso_saida
#> <chr> <int> <int> <int> <int> <int> <dbl>
#> 1 ABQ 2013 7 22 2145 2007 98
#> 2 ACK 2013 7 23 1139 800 219
#> 3 ALB 2013 1 25 123 2000 323
#> 4 ANC 2013 8 17 1740 1625 75
#> 5 ATL 2013 7 22 2257 759 898
#> 6 AUS 2013 7 10 2056 1505 351
#> # ℹ 102 more rows
#> # ℹ 12 more variables: horario_chegada <int>, chegada_prevista <int>, …Perceba que existem 105 destinos, mas obtivemos 108 linhas aqui. O que aconteceu? slice_min() e slice_max() mantém os valores empatados, então n = 1 significa “nos dê todas as linhas com o maior valor”. Se você quiser exatamente uma linha por grupo, você pode definir with_ties = FALSE.
Isso é similar a calcular o atraso máximo com summarize(), mas você obtém toda a linha correspondente (ou linhas, se houver um empate) em vez de apenas uma síntese estatística.
3.5.4 Agrupando por múltiplas variáveis
Você pode criar grupos utilizando mais de uma variável. Por exemplo, podemos fazer um grupo para cada data.
por_dia <- voos |>
group_by(ano, mes, dia)
por_dia
#> # A tibble: 336,776 × 19
#> # Groups: ano, mes, dia [365]
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Quando você cria um sumário de um tibble agrupado por mais de uma variável, cada sumário remove a camada do último grupo. Na verdade, essa não era uma boa forma de fazer essa função funcionar, mas é difícil mudar agora sem quebrar códigos que já existem. Para tornar óbvio o que está acontecendo, o dplyr exibe uma mensagem que te diz como modificar esse comportamento:
Se você acha que esse comportamente está adequado, você pode requisitá-lo explicitamente para que a mensagem seja suprimida:
Ou então, modifique o comportamento padrão definindo um valor diferente, por exemplo, "drop", para descartar todos os agrupamentos ou "keep" para manter os mesmos grupos.
3.5.5 Desagrupando
Você também pode querer remover agrupamentos de um data frame sem utilizar summarize(). Você pode fazer isso com ungroup().
por_dia |>
ungroup()
#> # A tibble: 336,776 × 19
#> ano mes dia horario_saida saida_programada atraso_saida
#> <int> <int> <int> <int> <int> <dbl>
#> 1 2013 1 1 517 515 2
#> 2 2013 1 1 533 529 4
#> 3 2013 1 1 542 540 2
#> 4 2013 1 1 544 545 -1
#> 5 2013 1 1 554 600 -6
#> 6 2013 1 1 554 558 -4
#> # ℹ 336,770 more rows
#> # ℹ 13 more variables: horario_chegada <int>, chegada_prevista <int>, …Agora, vamos ver o que acontece quando você tenta sumarizar um data frame desagrupado.
Você obtém uma única linha, pois o dplyr trata todas as linhas de um data frame desagrupado como pertencentes a um único grupo.
3.5.6 .by
dplyr 1.1.0 inclui uma nova e experimental sintaxe para agrupamentos por operação, trata-se do argumento .by. group_by() e ungroup() não serão abandonados, mas você pode também utilizar .by para agrupar no âmbito de uma única operação:
Ou, se você quiser agrupar por múltiplas variáveis:
.by funciona com todos os verbos e tem a vantagem de você não precisar utilizar o argumento .groups para suprimir a mensagem de agrupamento ou ungroup() quando já tiver terminado a operação.
Nós não focamos nessa sintaxe nesse capítulo pois ela era muito nova quando escrevemos o livro. No entanto, quisemos mencioná-la pois achamos que ela tem muito potencial e provavelmente se tonará bastante popular. Você pode ler mais sobre ela em dplyr 1.1.0 blog post.
3.5.7 Exercícios
Qual companhia aérea (companhia_aerea) possui a pior média de atrasos? Desafio: você consegue desvendar os efeitos de aeroportos ruins versus companhias aéreas ruins? Por que sim ou por que não? (Dica: experimente usar
voos |> group_by(companhia_aerea, destino) |> summarize(n()))Ache os vôos que estão mais atrasados no momento da decolagem, a partir de cada destino.
Como os atrasos variam ao longo do dia. Ilustre sua resposta com um gráfico.
O que acontece se você passar um
nnegativo paraslice_min()e funções similares?Explique o que
count()faz em termos dos verbos dplyr que você acabou de aprender. O que o argumentosortfaz para a funçãocount()?-
Suponha que temos o pequeno data frame a seguir:
-
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que
group_by()faz:df |> group_by(y) -
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que
arrange()faz. Comente também a respeito da diferença em relação aogroup_by()da parte (a):df |> arrange(y) -
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que o seguinte pipeline faz:
-
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que o seguinte pipeline faz. Em seguida, comente sobre o que a mensagem diz:
-
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que o seguinte pipeline faz. Como a saída difere da saída da parte (d):
-
Escreva como você acha que será a saída, e em seguida confira se acertou e descreva o que cada pipeline faz: Em que os dois pipelines diferem?
-
3.6 Estudo de caso: agregados e tamanho de amostra
Sempre que você fizer alguma agregação, é sempre uma boa ideia incluir a contagem (n()). Dessa foma, você pode se certificar que não está tirando conclusões baseadas em pequenas quantidades de dados. Vamos demonstrar isso com alguns dados de baseball do pacote dados. Especificamente, iremos comparar qual a proporção de vezes um jogador acertou a bola (“hit”) (rebatidas_simples) vs. o número de vezes que ele tentou colocar a bola em jogo (vez_bastao):
rebatedores <- dados::rebatedores |>
group_by(id_jogador) |>
summarize(
performance = sum(rebatidas_simples, na.rm = TRUE) / sum(vez_bastao, na.rm = TRUE),
n = sum(vez_bastao, na.rm = TRUE)
)
rebatedores
#> # A tibble: 20,730 × 3
#> id_jogador performance n
#> <chr> <dbl> <int>
#> 1 aardsda01 0 4
#> 2 aaronha01 0.305 12364
#> 3 aaronto01 0.229 944
#> 4 aasedo01 0 5
#> 5 abadan01 0.0952 21
#> 6 abadfe01 0.111 9
#> # ℹ 20,724 more rowsQuando montamos um gráfico da habilidade dos rebatedores (medidas pela média de acerto, performance) contra o número de oportunidades de acertar a bola (medidas pelo número de vezes ao bastão, n), é possível ver dois padrões:
A variação em
performanceé maior entre os jogadores que foram menos vezes ao bastão. A forma do gráfico é muito característica: sempre que você projetar no gráfico uma média (ou alguma outra sumarização estatística) vs. tamanho do grupo, você verá que a variação diminui à medida que o tamanho da amostra aumenta7.Existe uma correlação positiva entre a habilidade (
performance) e as oportunidades de acertar a bola (n), pois os times querem dar aos melhores jogadores o maior número de oportunidades de acertar a bola.
rebatedores |>
filter(n > 100) |>
ggplot(aes(x = n, y = performance)) +
geom_point(alpha = 1 / 10) +
geom_smooth(se = FALSE)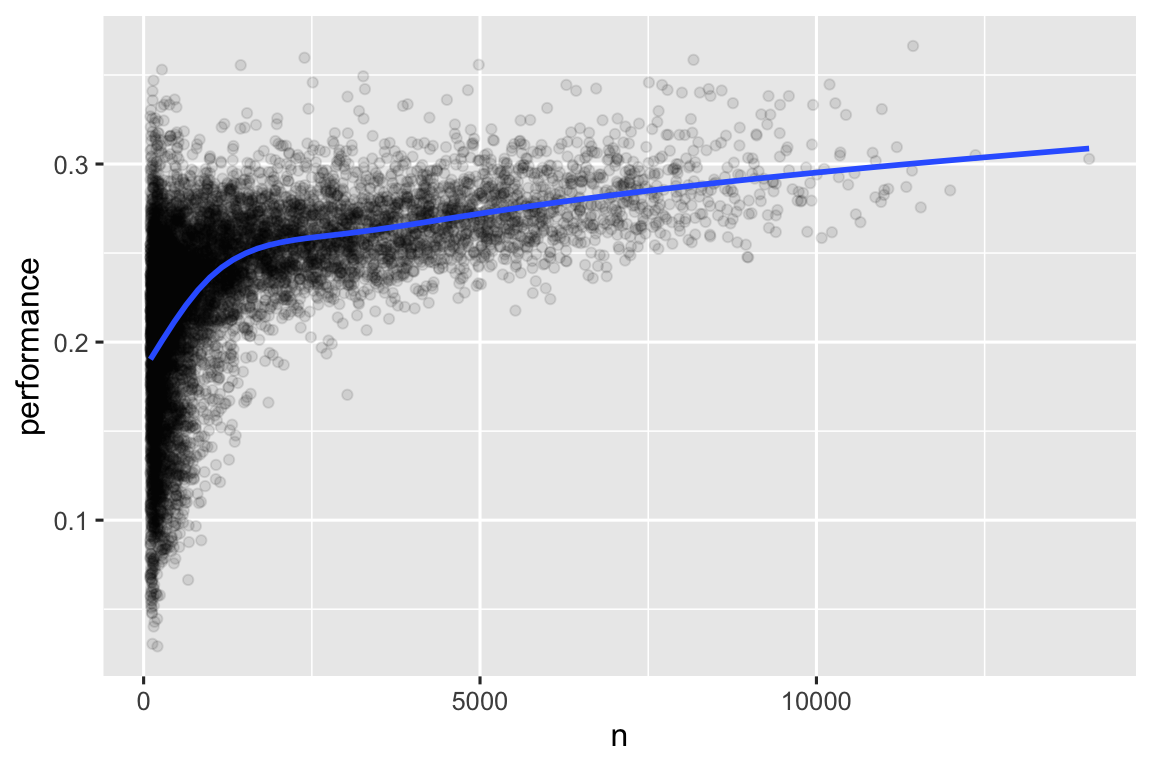
Perceba o padrão conveniente de combinar ggplot2 e dplyr. Você só precisa lembrar de mudar do |>, para processamento do conjunto de dados, para o + para adicionar camadas ao seu gráfico.
Isso também tem implicações importantes para ranqueamento. Se você simplesmente ordenar com desc(performance), as pessoas com as melhoras médias de rebatidas serão claramente aquelas que tentaram botar a bola em jogo apenas algumas poucas vezes mas calhou de acertarem, esses não são necessariamente os jogadores mais hábeis:
Você pode achar uma boa explicação sobre esse problema e como resolvê-lo em http://varianceexplained.org/r/empirical_bayes_baseball/ e https://www.evanmiller.org/how-not-to-sort-by-average-rating.html.
3.7 Resumo
Nesse capítulo, você aprendeu a usar as ferramentas que o pacote dplyr fornece para trabalhar com data frames.
Podemos considerar que elas estão agrupadas em três categorias: as que manipulam as linhas (como filter() e arrange(), as que manipulam colunas (como select() e mutate()), e aquelas que manipulam grupos (como group_by() e summarize()). Nesse capítulo, nós focamos nas ferramentas que trabalham “no data frame como um todo”, mas você ainda não aprendeu muito sobre o que é possível fazer com uma variável individualmente. Nós voltaremos para essa assunto na parte do livro sobre Transformações, onde cada capítulo vai te mostrar ferramentas para cada tipo específico de variáveis.
No próximo capítulo, voltaremos ao fluxo de trabalho para discutir sobre a importância do estilo de código, mantendo seu código bem organizado facilitando que você e outros leiam e entendam.
Nota de tradução: No pacote dplyr, chamamos as principais funções de “verbos” para enfatizar que elas fazem uma ação. Os principais verbos do dplyr são:
filter()(filtrar),arrange()(ordenar),select()(selecionar),mutate()(alterar),summarize()(sumarizar), egroup_by()(agrupar).↩︎de double precision floating number - remete à forma como números reais são representados nos computadores, utilizando 64 bits em vez dos tradicionais 32 bits de um inteiro comum, por isso “double” (dobro).↩︎
Depos você irá aprender sobre a família de funções
slice_*()que permite escolher linhas com base em suas posições.↩︎no hemisfério norte↩︎
Lembre-se que no RStudio, o jeito mais fácil de visualizar um conjunto de dados com muitas colunas é
View().↩︎Ou
summarise(), se você preferir o inglês britânico.↩︎*cof-cof* a lei dos grandes números *cof-cof*.↩︎